Fosforylacja oksydacyjna
Fosforylacja oksydacyjna – szlak metaboliczny, w którego wyniku energia, uwalniana podczas utleniania zredukowanych nukleotydów, przekształcana jest w energię ATP. Organizmy żywe wykorzystują wiele różnych związków organicznych, jednak aby wytworzyć z nich energię przydatną metabolicznie, cząsteczki ATP, w większości przeprowadzają fosforylację oksydacyjną. Szlak ten jest dominujący ze względu na wysoką efektywność w porównaniu do alternatywnych sposobów syntezy ATP, czyli fermentacji.
Podczas fosforylacji oksydacyjnej, w wyniku szeregu reakcji redoks, elektrony przenoszone są ze zredukowanych nukleotydów, NADH i FADH2, na pełniący funkcję akceptora elektronów tlen. Zachodzące reakcje prowadzą do zmagazynowania energii, służącej następnie do syntezy ATP. W komórkach eukariotycznych szereg reakcji redoks zachodzi na kompleksach białkowych znajdujących się w mitochondriach. W komórkach prokariotycznych kompleksy białkowe zlokalizowane są w błonach komórkowych. Zestaw enzymów biorących udział w przenoszeniu elektronów określa się jako łańcuch oddechowy. U eukariotów składa się on z pięciu głównych enzymów, u prokariotów odnaleziono wiele różnych enzymów pełniących funkcję donorów i akceptorów elektronów.
Energia uwalniana podczas transportu elektronów w łańcuchu oddechowym zużywana jest do przenoszenia protonów przez wewnętrzną błonę mitochondrialną. Proces ten przez jego odkrywcę został nazwany chemiosmozą. Energia potencjalna gromadzona jest w postaci gradientu pH i potencjału elektrycznego w poprzek błony. Zgromadzona w tej formie energia wykorzystywana jest przez kompleks enzymatyczny syntazy ATP, który pozwala protonom przejść przez błonę zgodnie z gradientem stężeń. Enzym ten zamienia jednocześnie energię gradientu pH i elektrycznego na energię wiązań chemicznych ATP, wytwarzanego przez przyłączenie do ADP reszty kwasu ortofosforowego, czyli reakcji fosforylacji. Niezwykłość reakcji syntezy ATP związana jest z obracaniem się części enzymu napędzanej przepływającymi protonami, przypominając działanie silnika elektrycznego. Obrót części enzymu odłącza wytworzoną cząsteczkę ATP.
Fosforylacja oksydacyjna jest ważnym procesem metabolicznym, jednak jej zachodzenie prowadzi do powstawania reaktywnych form tlenu, takich jak nadtlenek wodoru oraz wolnych rodników, niszczących komórki, a w efekcie powodujących choroby i prawdopodobnie przyspieszających starzenie się. Enzymy przeprowadzające ten szlak metaboliczny są wrażliwe na wiele leków i trucizn, takich jak cyjanek.
Wstęp
Fosforylacja oksydacyjna zachodzi dzięki dostarczaniu energii w szeregu reakcji określanych jako łańcuch oddechowy i zużywaniu zmagazynowanej energii w reakcji przeprowadzanej przez syntazę ATP. Pierwsza i druga reakcja są ze sobą sprzężone. Oznacza to, że żadna nie może zachodzić bez zachodzenia drugiej. Przepływ elektronów z donorów w postaci cząsteczek NADH na akceptory w postaci cząsteczek tlenu, odbywający się przez szereg przenośników biorących udział w łańcuchu transportu elektronów, jest procesem egzoenergetycznym – uwalniającym energię. Synteza ATP jest zaś procesem endoenergetycznym który, aby zachodzić, wymaga dostarczenia energii. Zarówno łańcuch oddechowy, jak i synteza ATP zachodzi na błonach białkowo-lipidowych. Energia z łańcucha transportu elektronów jest przenoszona na syntazę ATP, dzięki wytworzeniu różnicy stężeń jonów w poprzek błony, nazywanej gradientem elektrochemicznym. Proces przenoszenia protonów przez błonę został nazwany chemiosmozą[1]. Przenoszenie protonów przez błonę odbywa się dzięki enzymom obecnym w błonach, tłoczącym protony z jednej strony na drugą podczas zachodzenia łańcucha transportu elektronów i określanych nazwą pompy protonowe. Enzymy te podobnie jak urządzenia elektryczne wykonują pracę, gdy przepływa przez nie prąd elektryczny. Powstający, w efekcie ich pracy, gradient elektrochemiczny, nazywany często siłą protonomotoryczną, składa się z dwóch elementów: różnicy stężeń protonów (gradient pH) oraz różnicy potencjałów, wynikającej z ładunków przemieszczanych cząsteczek (po jednej stronie błony istnieje ładunek ujemny, po drugiej dodatni). W przypadku mitochondriów większe znaczenie odgrywa różnica potencjałów, a w przypadku chloroplastów większe znaczenie ma gradient pH[2].
Syntaza ATP zużywa energię gradientu elektrochemicznego, pozwalając przejść ładunkom (protonom) z powrotem przez błonę[3]. Enzym ten działa podobnie jak silnik elektryczny, wykorzystując energię w postaci siły protonomotorycznej do obracania jednym z białek kompleksu. Obroty umożliwiają wytworzenie ATP.
Ilość energii wytworzona podczas fosforylacji oksydacyjnej jest zdecydowanie większa od ilości produkowanych podczas zachodzenia fermentacji. Glikoliza dostarcza jedynie dwie cząsteczki ATP, podczas gdy w procesie fosforylacji oksydacyjnej z 10 cząsteczek NADH i dwie bursztynianu, powstałych przy pełnym utlenieniu cząsteczki glukozy do wody i dwutlenku węgla powstaje około 30–36 cząsteczek ATP[4]. Podana ilość ATP jest maksymalną teoretyczną wydajnością, w rzeczywistości cześć protonów przenika przez błonę, omijając syntazę ATP i obniżając wydajność zamiany gradientu elektrochemicznego na ATP[5].
Cząsteczki przenoszące elektrony i protony
Zachodzenia łańcucha oddechowego prowadzi do przemieszczania są zarówno protonów, jak i elektronów. Podczas przemieszczania elektronów z donorów na akceptory następuje przeniesienie protonów przez błonę białkowo-lipidową. W procesie uczestniczą związki budujące błony oraz cząsteczki rozpuszczalne. W mitochondrialnym łańcuchu oddechowym elektrony przenoszone są między innymi w przestrzeni międzybłonowej przez niewielkie białko rozpuszczalne, cytochrom c[6]. Cząsteczka ta przenosi jedynie elektrony, dzięki redukcji i utlenianiu atomów żelaza zawartego w hemie, będącego składnikiem cytochromu. Cytochrom c został także wykryty w przestrzeni peryplazmatycznej części bakterii[7].
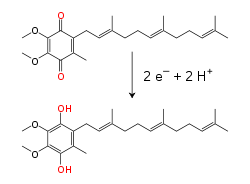
W wewnętrznej błonie mitochondrialnej lipofilny przenośnik elektronów, koenzym Q10 (Q), przenosi zarówno elektrony, jak protony, dzięki cyklicznej redukcji i utlenianiu[8]. Mała cząsteczka benzochinonu, o silnie hydrofobowym charakterze może swobodnie dyfundować w błonie. Po przyjęciu dwóch elektronów i dwóch protonów ubichinon przekształca się w formę zredukowaną – ubichinol (QH2). Gdy QH2 podczas utleniania przekazuje dwa elektrony i uwalnia dwa protony, powraca do swojej formy utlenionej – ubichononu. W efekcie, dzięki lokalizacji miejsca redukcji Q po jednej stronie błony, a miejsca utleniania QH2 po drugiej, ubichinon bierze udział w przenoszeniu protonów przez błonę[9]. U części bakterii poza ubichinonem w łańcuchu transportu elektronów uczestniczą inne chinony, na przykład menachinon[10].
Białka uczestniczące w łańcuchu oddechowym przenoszą elektrony dzięki obecności grup flawinowych[11][3], centrów żelazo-siarkowych i cytochromów. Istnieje kilka typów centrów żelazo-siarka. Najprostsze, ze znalezionych w łańcuchu oddechowym, zawiera dwa atomu żelaza i dwa atomu nieorganicznej siarki i jest określane jako centrum [2Fe–2S]. Drugi rodzaj, nazywany [4Fe–4S], zawiera sześcian zbudowany z czterech atomów żelaza i czterech atomów siarki. Każdy z atomów żelaza wchodzących w skład centrów koordynowany jest przez aminokwas, zwykle grupę -SH cysteiny. Jony metalu, tworzącego kofaktor, ulegają redukcji podczas przenoszenia elektronów przez przenośniki białkowe. W tym przypadku wraz z elektronem nie są przemieszczane protony. Elektrony mogą pokonywać stosunkowo duże odległości podczas przemieszczania się przez szereg kofaktorów[12]. Następuje to dzięki zjawisku tunelowemu, które umożliwia szybkie pokonanie odległości mniejszych niż 1,4 10−9 m[13].
Łańcuch transportu elektronów u eukariotów
Wiele katabolicznych szlaków metabolicznych, takich jak glikoliza, cykl kwasu cytrynowego, β-oksydacja, prowadzi do wytworzenia NADH. Koenzym ten zawiera elektrony, które mają wysoki potencjał standardowy, prowadzą do uwolnienia dużej ilości energii podczas utleniania. Komórka nie uwalnia całej tej energii natychmiast, w sposób niekontrolowany. Elektrony są pobrane z NADH przeniesione na tlen przez kilka enzymów, których każdy uwalnia tylko cześć energii. Ten zestaw enzymów, zawierający kompleksy od I do IV, nazywany jest łańcuchem transportu elektronów i znajduje się w wewnętrznej błonie mitochondrialnej. W reakcjach łańcucha oddechowego utleniany jest także bursztynian, jednak wchodzi do szlaku w dodatkowym punkcie.
W komórkach eukariotycznych, enzymy przenoszące elektrony, wykorzystują energię uwalnianą podczas utleniania NADH do przeniesienia protonów przez wewnętrzną błonę mitochondrialną do przestrzeni międzybłonowej i wytwarzają gradient elektrochemiczny w poprzek błony. Energia zmagazynowana w postaci gradientu zużywana jest przez syntazę ATP do wytwarzania ATP. Fosforylacja oksydacyjna w mitochondriach eukariotów jest najlepiej poznanym przykładem tego procesu. Mitochondria są obecne u prawie wszystkich eukariotów z wyjątkiem oddychających beztlenowo pierwotniaków takich jak Trichomonas vaginalis oraz niektórych grzybów, u których protony przekształcane są do wodoru w zmodyfikowanych mitochondriach określanych jako hydrogenosomy[14][15].
Oksydoreduktaza NADH-koenzym Q (Kompleks I)

Oksydoreduktaza NADH-koenzym Q, nazywana także dehydrogenazą NADH lub kompleksem I, jest pierwszym białkiem łańcucha transportu elektronów[16]. U ssaków kompleks I jest ogromnym enzymem, składającym się z 46 podjednostek o łącznej masie około 1000 kDa[17]. Struktura kompleksu została dobrze poznana jedynie u bakterii[18], u większości organizmów kompleks ma kształt litery L z poziomym ramieniem umieszczonym w błonie a pionowym skierowanym do wnętrza mitochondrium[19][20]. Geny kodujące poszczególne podjednostki, podobnie jak ma to miejsce dla wielu enzymów mitochondrialnych, zawarte są zarówno w jądrze komórkowym, jak i genomie mitochondrialnym.
Reakcja katalizowana przez enzym polega na przeniesieniu dwóch elektronów z NADH na koenzym Q10 (ubichinon, Q), związek lipofilny swobodnie dyfundujący w błonie mitochondrialnej:
Reakcja rozpoczynająca łańcuch transportu elektronów rozpoczyna się od przyłączenia cząsteczki NADH do kompleksu I i oderwaniu dwóch elektronów. Elektrony przekazywane są na kompleks przez grupę prostetyczną wchodzącą w skład enzymu, mononukleotyd flawinowy (FMN). Przekazanie dwóch elektronów na FMN przekształca go w zredukowaną formę, FMNH2. Następnie elektrony przenoszone są przez kolejne centra żelazo-siarka, drugi rodzaj grup prostetycznych obecnych w kompleksie[18]. W enzymie znajdują się centra [2Fe-2S] oraz [4Fe-4S].
Podczas przejścia przez kompleks dwóch elektronów cztery protony przemieszczane są z macierzy mitochondrialnej do przestrzeni międzybłonowej. Mechanizm przenoszenia protonów nie jest w pełni poznany, prawdopodobnie dochodzi do zmian konformacyjnych, w których wyniku protony przyłączone po stronie macierzy mitochondrialnej zostają przeniesione na stronę przestrzeni międzybłonowej i tam odłączone od białka[21]. Pobrane elektrony poprzez centra żelazo-siarka przenoszone są ostatecznie na cząsteczkę ubichinonu w błonie[16]. Zredukowanie ubichinonu również przyczynia się do wytworzenia gradientu protonowego, ponieważ oba przyłączane protony pobierane są z macierzy mitochondrialnej, co prowadzi do wytworzenia ubichonolu (QH2).
Oksydoreduktaza bursztynian-ubichinon (kompleks II)
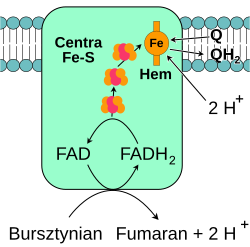
Oksydoreduktaza bursztynian-ubichinon, określana także jako kompleks II lub dehydrogenaza bursztynianowa, jest drugim punktem wejścia elektronów do łańcucha transportu elektronów[22]. Jest to szczególny kompleks, który jest zarówno enzymem cyklu kwasu cytrynowego, jak i łańcucha oddechowego. Kompleks II składa się z czterech podjednostek oraz zawiera kofaktor w postaci dinukleotydu flawinoadeninowego (FAD), centra żalazo-siarka oraz grupę hemową, która nie uczestniczy w przenoszeniu elektronów na koenzym Q, lecz prawdopodobnie odgrywa rolę w zmniejszeniu produkcji reaktywnych form tlenu[23][24]. Enzym utlenia bursztynian do fumaranu, redukując jednocześnie ubichinon. W reakcji tej uwalniane jest mniej energii niż podczas utleniania NADH, nie dochodzi również do przemieszczania protonów przez wewnętrzną błonę mitochondrialną. Kompleks II nie bierze więc udziału w wytwarzaniu gradientu protonowego.
U niektórych eukariotów, takich jak należąca do pasożytniczych nicieni Ascaris suum, enzym zbliżony do kompleksu II, reduktaza fumaranu (oksydoreduktaza menachinon:fumaran, QFR – ang. menaquinol:fumarate oxidoreductase), przeprowadza reakcję odwrotną, redukując fumaran. Pozwala to przeżyć pasożytowi w warunkach beztlenowych panujących w jelicie grubym, poprzez przeprowadzanie beztlenowej fosforylacji oksydacyjnej, z fumaranem jako akceptorem elektronów[25]. Innym przykładem nietypowego wykorzystania kompleksu II jest pasożytniczy pierwotniak wywołujący malarię Plasmodium falciparum. U tego organizmu odwrócenie działania kompleksu II umożliwi odtwarzanie ubichinonu, który jest następnie zużywany w wyjątkowym procesie biosyntezy pirymidyn[26].
Oksydoreduktaza flawoproteina przenosząca elektron-ubichinon
Oksydoreduktaza flawoproteina przenosząca elektrony-ubichinon (oksydoreduktaza ETF-Q), nazywana także dehydrogenazą flawoproteiny przenoszącej elektrony, jest trzecim punktem wejścia do łańcucha transportu elektronów. Jest to enzym odbierający elektrony od flawoproteiny przenoszącej elektrony występującej w macierzy mitochondrialnej i redukujący pulę ubichinonu w błonie[27]. Enzym zawiera flawinę i centrum żelazo-siarka typu [4Fe-4S], jednak w przeciwieństwie do innych kompleksów nie jest białkiem transbłonowym, lecz pozostaje związany z wewnętrzną powierzchnią błony[28].
U ssaków enzym odgrywa ważną rolę w β-oksydacji kwasów tłuszczowych, katabolizmie aminokwasów i choliny, spełniając funkcję akceptora elektronów dehydrogenazy acylo-CoA[29][30]. U roślin, oksydoreduktaza ETF-Q nie bierze udziału w β-oksydacji, jednak odgrywa ważną rolę w metabolizmie podczas długich okresów ciemności[31].
Oksydoreduktaza koenzym Q-cytochrom c (kompleks III)
Oksydoreduktaza koenzym Q-cytochrom c nazywana także reduktazą cytochromu c, kompleksem cytochromów bc1 lub kompleksem III[32][33]. U ssaków enzym składa się z dwóch identycznych podjednostek, z których każda składa się z 11 podjednostek białkowych, centrum żelazo-siarka [2Fe-2S] i trzech cytochromów: cytochromu c1 i dwóch cytochromów b[34]. Cytochromy są białkami przenoszącymi elektrony, zawierającymi jedną lub więcej grup hemowych. Atomy żelaza obecne w kompleksie III znajdują się na przemian na +2 lub +3 stopniu utlenienia, przyjmując lub oddając elektron.
Reakcja katalizowana przez kompleks III polega na utlenieniu cząsteczki ubichinolu i redukcji dwóch cząsteczek cytochromu c, białka zawierającego hem, luźno związanego z błoną mitochondrialną. W przeciwieństwie do cząsteczki koenzymu Q, przenoszącej dwa elektrony, cytochrom c przenosi tylko jeden elektron.
Ze względu na możliwość przeniesienia tylko jednego elektronu z cząsteczki QH2 na cytochrom c, reakcje zachodzące na kompleksie III są bardziej złożone niż reakcje na pozostałych kompleksach i zachodzą w dwóch etapach nazywanych cyklem Q[35]. W pierwszym etapie z kompleksem związane są trzy substraty, utleniana cząsteczka QH2, cytochrom c oraz cząsteczka ubichinonu. Jeden z elektronów pobranych z ubichinolu przekazywany jest na cytochrom c. Drugi elektron przekazywany jest do cząsteczki ubichinonu przyłączonej po stronie macierzy mitochondrialnej. Protony powstające po pobraniu elektronów z cząsteczki QH2 uwalniane są do przestrzeni międzybłonowej. Cząsteczka ubichinonu redukowana po przeciwnej stronie kompleksu przyłącza protony z macierzy mitochondrialnej. Po odebraniu elektronów z ubichinolu odłącza się on od kompleksu. To samo dzieje się ze zredukowanym cytochromem c. Druga cząsteczka ubichinonu, na którą został przeniesiony jeden elektron, tworzy semichinon i pozostaje związana z kompleksem. W drugim etapie do enzymu przyłączona zostaje następna cząsteczka QH2 i po raz kolejny jeden z elektronów przenoszony jest na cytochrom c a drugi na związaną cząsteczkę semichinonu. W efekcie semichinon zostaje zredukowany do QH2, a dwa dodatkowe protony pobrane z macierzy mitochondrialnej. Powstała cząsteczka QH2 odrywa się od kompleksu i może zostać utleniona w części utleniającej enzymu[36].
Utlenianie koenzymu Q do ubichinonu po zewnętrznej stronie błony i redukcja do ubichinolu po stronie wewnętrznej daje efekt przenoszenia protonów przez błonę, co prowadzi do zwiększenia gradientu elektrochemicznego[3]. Dwa etapy utleniania ubichinolu zwiększają efektywność przenoszenia protonów. Bez zachodzenia cyklu Q redukcji jednej cząsteczki cytochromu c towarzyszyłoby przeniesienie przez błonę tylko jednego protonu[3].
Oksydaza cytochromu c (Kompleks IV)

Oksydaza cytochromu c, określana także jako kompleks IV, jest ostatnim enzymem łańcucha transportu elektronów[37]. Kompleks IV mitochondriów ssaków zbudowany jest z 13 podjednostek, dwóch cząsteczek hemu oraz wielu kofaktorów w postaci jonów metali – trzech atomów miedzi, jednego magnezu i jednego cynku[38].
Enzym katalizuje końcową reakcje łańcucha oddechowego, przenosząc elektrony na tlen i jednocześnie przemieszczając protony przez błonę[39]. Tlen po przyjęciu elektronów ulega redukcji, przyłączając protony z macierzy mitochondrialnej, co prowadzi do powstania wody. Zarówno przenoszenie protonów w poprzek błony, jak i zużywanie, po stronie macierzy mitochondrialnej w reakcji powstawania wody, prowadzi do zwiększenia gradientu protonowego. Reakcja katalizowana polega na utlenianiu cytochromu c i redukcji tlenu według równania:
Alternatywne reduktazy i oksydazy
U wiele organizmów eukariotycznych w łańcuchu transportu elektronów mogą brać także enzymy nieopisane powyżej. W mitochondriach roślin występują dodatkowe enzymy, które mogą utleniać cytozolowe NADH lub NADPH po stronie przestrzeni międzybłonowej i także przenosić elektrony na pulę ubichinonu[40]. Enzymy te nie przenoszą protonów przez błonę mitochondrialną, a tym samym nie biorą udziału w wytwarzaniu gradientu elektrochemicznego[41].
Innym przykładem odmiennego łańcucha transportu elektronów jest występowanie oksydazy alternatywnej, która występuje w komórkach roślin, części grzybów, protistów i prawdopodobnie niektórych zwierząt[42][43] Enzym ten przenosi elektrony bezpośrednio z ubichinolu na tlen, z pominięciem kompleksów III i IV, nie wytwarzając gradientu elektochemicznego[44].
Transport elektronów z udziałem alternatywnych oksydaz NAD(P)H i ubichinolu w znikomym stopniu prowadzi do syntezy ATP w stosunku zwykłej drogi. Korzyści wynikające ze skróconego łańcucha oddechowego nie zostały do końca wyjaśnione. Wiadomo, jednak że oksydaza alternatywna produkowana jest w odpowiedzi na stres w postaci zimna, reaktywnych form tlenu, infekcji patogenów i innych czynników zmniejszających wydajność pełnego łańcucha transportu elektronów[45][46]. Alternatywne drogi przenoszenia elektronów mogą, zwiększać odporność organizmów na czynniki szkodliwe, poprzez zwalczanie stresu oksydacyjnego[47].
Organizacja kompleksów
Początkowy model ułożenia kompleksów łańcucha oddechowego zakładał swobodne i niezależne rozmieszczanie w wewnętrznej błonie mitochondrialnej[48]. Nowsze badania wskazują, że enzymy mogą tworzyć większe kompleksy określane jako superkompleksy lub „respirosomy”[49]. W tym modelu kompleksy zorganizowane są w zbiory wzajemnie oddziałujących enzymów[50]. Takie zespoły pozwalałyby przenosić substraty pomiędzy poszczególnymi kompleksami, zwiększając szybkość i wydajność transferu elektronów[51]. W superkompleksach występujących u ssaków niektóre elementy mogą być obecne w większych ilościach niż pozostałe, a stosunek pomiędzy kompleksami I/II/III/IV i syntazą ATP wynosiłby około 1:1:3:7:4[52]. Jednakże istnienie superkompleksów nie jest pewne, a cześć wyników nie potwierdza takiego modelu rozkładu kompleksów łańcucha oddechowego[17][53].
Łańcuch transportu elektronów u prokariotów
W przeciwieństwie do ogólnego podobieństwa w strukturze i działaniu łańcucha oddechowego w komórkach eukariotycznych, u bakterii i archeonów istnieje o wiele większa różnorodność enzymów biorących udział w przenoszeniu elektronów. Jako akceptor elektronów może być użyte wiele substancji chemicznych[54]. Podobnie jak u eukariotów transport elektronów przez kolejne przenośniki prowadzi do przenoszenia protonów przez błonę i wytwarzania gradientu elektrochemicznego. Przebieg fosforylacji oksydacyjnej został dobrze poznany na przykładzie Escherichia coli u bakterii, jednak u archeanów przebieg procesu jest stosunkowo mało poznany[55].
Główną różnicą w fosforylacji oksydacyjnej pomiędzy komórkami eukariotycznymi a prokariotycznymi jest używanie przez bakterie i archeany wielu różnych substancji jako donora lub ostatecznego akceptora elektronów. Pozwala to na wzrost organizmów prokariotycznych w wielu różnych warunkach środowiska[56]. Na przykład u E. coli, fosforylacja oksydacyjna może być napędzana przez liczne pary związków utlenianych i redukowanych, które zostały wymienione w tabeli. Potencjał redoks obrazuje ile energii uwalniane jest podczas utleniania lub redukcji, dla redukcji wartość potencjału jest ujemna, a dla utleniania wartości są dodatnie.
| Enzym oddechowy | Pary redoks | Potencjał redoks [Wolty] |
|---|---|---|
| Dehydrogenaza mrówczanu | Wodorowęglan/Mrówczany | –0,43 |
| Hydrogenaza | Proton/Wodór | –0,42 |
| Dehydrogenaza NADH | NAD+/NADH | –0,32 |
| Dedrogenaza glicerolo-3-fosforanowa | DHAP/Glicerolo-3-fosforan | –0,19 |
| Oksydaza pirogronianowa | Octan + Dwutlenek węgla/Pirogronian | ? |
| Dehydrogenaza mleczanowa | Pirogronian/Mleczan | –0,19 |
| Dehydrogenaza D-aminokwasów | 2-oksokwas + amoniak/D-aminokwasy | ? |
| Dehydrogenaza glukonianu | Glukonian/Glukoza | –0,14 |
| Dehydrogenaza bursztynianu | Fumaran/Bursztynian | +0,03 |
| Oksydaza ubichinolu | Tlen/Woda | +0,82 |
| Reduktaza azotanowa | Azotan/Azotyn | +0,42 |
| Reduktaza azotynowa | Azotyn/Amoniak | +0,36 |
| Reduktaza dimetylosulfotlenku | DMSO/DMS | +0,16 |
| Reduktaza tlenku trimetyloaminy | TMAO/TMA | +0,13 |
| Reduktaza fumaranu | Fumaran/Bursztynian | +0,03 |
Jak przedstawiono w tabeli, E. coli może do wytwarzania energii metabolicznej używać związki takie jak mrówczany, wodór, kwas mlekowy jako donory elektronów i azotany, DMSO lub tlen jako akceptory[56]. Wyższy potencjał redoks oznacza uwolnienie większej ilości energii podczas reakcji. Niezwykłą parę tworzą bursztynian/fumaran, przy których potencjał redoks jest niemal równa zeru. Dlatego też przy utlenianiu bursztynianu konieczny jest silny utleniacz w postaci tlenu lub fumaran może być zredukowany do bursztynianu przy użyciu silnego reduktora, czyli mrówczanu. Te alternatywne reakcje są katalizowane przez odpowiednio dehydrogenazę bursztynianu lub reduktazę fumaranu[58].
Niektóre prokarioty używają par związków o niskim potencjale redoks. Na przykład bakterie nitryfikacyjne, takie jak Nitrobacter, utleniają azotyny do azotanów, przekazując elektrony na tlen. Niewielka ilość energii uwalniana podczas reakcji wystarcza do przenoszenia protonów przez błonę i syntezy ATP, lecz nie wystarcza do wytworzenia NADH lub NADPH niezbędnych do procesów anabolicznych[59]. Ten problem zostaje rozwiązany dzięki oksydoreduktazie azotynowej, która wytwarza wystarczająco duża siłę protonomotoryczną, aby wymusić odwrotny przebieg łańcucha transportu elektronów, powodując wytwarzanie NADH[60][61].
Użycie odpowiednich donorów lub akceptorów elektronów przez prokarioty regulowane jest przez czynniki środowiskowe[62]. Elastyczność procesów metabolicznych jest możliwa dzięki używaniu przez wielu oksydaz i reduktaz tej samej pulu ubichinonu. Pozwala to wielu zestawom enzymów współdziałać, poprzez współdzielenie ubichinonu obecnego w błonach jako związku pośredniego[57]. Łańcuch oddechowy o strukturze modułowej umożliwia łatwą zmianę zestawów enzymów.
Poza metaboliczną różnorodnością prokarioty posiadają także zestaw izoenzymów – różnych enzymów, katalizujących te same reakcje. Na przykład u E. coli występują dwie różne oksydazy ubichinonu, używające tlenu jako akceptora elektronów. W warunkach dobrej dostępności tlenu komórka używa oksydazy o niskim powinowactwie do tlenu, która jest zdolna do przeniesienia dwóch protonów na każdy elektron. Gdy poziom tlenu spadnie, komórka przenosi elektrony na tlen poprzez oksydazę, która nie bierze udziału w wytwarzaniu gradientu elektrochemicznego, jednak posiada wysokie powinowactwo do tlenu[63].
Syntaza ATP
Syntaza ATP, nazywana także kompleksem V, jest ostatnim enzymem biorącym udział w szeregu reakcji fosforylacji oksydacyjnej. Enzym ten został znaleziony we wszystkich żywych organizmach, zarówno prokariotycznych, jak i eukariotycznych[64]. Enzym zużywa energię zgromadzoną w postaci gradientu elektrochemicznego do syntezy ATP z ADP i fosforanu nieorganicznego (Pi). Do syntezy jednej cząsteczki ATP potrzebne jest przejście przez syntazę od 3 do 4 protonów[65][66], w niektórych komórkach ilość protonów może się zmieniać, w zależności od warunków[67].
Reakcja fosforylacji jest reakcją odwracalną, a przewaga jednego z kierunków reakcji zależy od siły protonomotorycznej. Przy braku gradientu protonowego, syntaza ATP przeprowadza reakcję hydrolizy ATP, przenosząc jednocześnie protony przez błonę. Gdy jednak siła protonomotoryczna jest odpowiednio duża, zachodzi odwrotna reakcja, protony przepływają zgodnie z gradientem stężeń, a ADP łączone jest z Pi z wytworzeniem ATP[64]. Bardzo podobny enzym H+-ATPaza obecna w błonie wakuoli, wywołuje obniżenie pH wnętrza organellum, przenosząc protony i jednocześnie hydrolizując ATP[68].
Syntaza ATP jest dużym kompleksem, kształtem przypominającym grzyb. U ssaków kompleks syntazy składa się z 16 podjednostek o łącznej masie około 600 kDa[69]. Domena będąca białkiem błonowym określana jest nazwą Fo i zawiera pierścień zbudowany z podjednostek c oraz kanał jonowy. Trzon wraz z przytwierdzoną do niego częścią kulistą, określany jest nazwą F1, jest miejscem syntezy ATP. Domena F1 składa się z sześciu podjednostek, należących do dwóch grup: trzech podjednostek α i trzech podjednostek β. Trzon łączący część kulista z domeną błonową zawiera tylko jedną podjednostkę γ[70]. Podjednostki α i β wiążą nukleotydy, jednak tylko podjednostka β katalizuje reakcję syntezy ATP. Wystająca z domeny F1, skierowany w stronę błony, to przypominająca pręt podjednostka kotwicząca zespół podjednostek α i β w głównej części kompleksu.
Protony przepływając przez kanał jonowy domeny Fo, powodują obracanie się zestawu podjednostek c[71]. Obroty prawdopodobnie powodowane są zmianami w jonizacji aminokwasów w pierścieniu podjednostek c, co wywołuje zmiany elektrostatyczne, które napędzają pierścień podjednostek c przy kanale jonowym[72]. Obroty pierścienia przenoszone są przez, tworzącą oś, podjednostkę γ do wnętrza podjednostek α i β, które nie mogą się obracać ze względu na tworzącą stojan długa podjednostkę w kształcie pręta. Ruch obrotowy podjednostki γ wewnątrz kulistej struktury podjednostek α i β dostarcza energii do centrów aktywnych na podjednostkach β, powodując oderwanie cząsteczek ATP[73].
Reakcja syntezy ATP zachodzi dzięki mechanizmowi zmian konformacyjnych, centrum aktywne podjednostki β występuje w dwóch cyklicznie zmieniających się stanach[74]. W stanie pierwszym jest otwarte i może przyłączyć cząsteczkę ADP i Pi. Gdy oba substraty zostaną przyłączone, białko zmienia kształt, powodując połączenie cząsteczek. Powstała cząsteczka ATP ma wysokie powinowactwo do enzymu i jest z nim silnie związana. Ostatni etap polega na powrocie enzymu do stanu pierwszego, centrum aktywne otwiera się, uwalniając cząsteczkę ATP i pozostając otwarte dla kolejnych substratów. Synteza ATP nie wymaga dostarczenia energii. Energia obrotów podjednostki γ konieczna jest do oderwania wytworzonej cząsteczki ATP (otwarcia centrum aktywnego).
U części bakterii i archeanów, synteza ATP napędzana jest nie przepływem protonów przez błonę komórkową, lecz jonów sodowych[75][76]. Archenany takie jak Methanococcus posiadają także syntazę A1Ao, formę enzymu o niewielkim podobieństwie sekwencji aminokwasów do białek syntazy ATP obecnych u pozostałych bakterii i komórek eukariotycznych. Prawdopodobnie u części gatunków forma syntazy A1Ao napędzana jest jonami sodu[77], jednak jest możliwe, że jest tak u wszystkich organizmów z tą postacią enzymu[76].
Reaktywne formy tlenu
Tlen cząsteczkowy, jako silny utleniacz, jest idealnym końcowym akceptorem elektronów. Redukcja tlenu może jednak powodować powstawanie szkodliwych związków pośrednich[78]. Chociaż przeniesienie czterech elektronów połączone z przyłączeniem czterech protonów prowadzi do powstania wody, która jest całkowicie nieszkodliwa, przeniesienie jednego lub dwóch elektronów prowadzi do powstania, szczególnie reaktywnych jonów ponadtlenkowych lub nadtlenkowych.
Reaktywne formy tlenu i produkty ich reakcji, takie jak rodnik hydroksylowy, są bardzo szkodliwe dla komórek, ponieważ powodują utlenianie białek, lipidów i wywołują mutacje w DNA. Uszkodzenia komórek prowadza do chorób i są jedną z przyczyn starzenia się[79][80].
Kompleks oksydazy cytochromowej bardzo efektywnie przeprowadza redukcję tlenu do wody i produkuje wyjątkowo mało częściowo zredukowanych związków pośrednich, jednak niewielkie ilości jonów ponadtenkowych i nadtlenkowych są wytwarzane podczas działania łańcucha oddechowego[81]. Szczególnie ważną reakcją jest redukcja koenzymu Q na kompleksie III, gdzie jako związek pośredni powstaje, wysoce reaktywny, semiubichinon. Ten niestabilny związek może prowadzić do „wycieku” elektronów, gdy zostaną one przeniesione bezpośrednio na tlen, tworząc nadtlenek[82].
W celu neutralizacji reaktywnych form tlenu komórki posiadają systemy licznych przeciwutleniaczy, w tym witaminy C i E, a także enzymy rozkładające związki szkodliwe chroniąc komórkę przed uszkodzeniem. Do enzymów chroniących komórkę przed reaktywnymi formami tlenu należą: dysmutaza ponadtlenkowa, katalaza i peroksydazy[78].
Inhibitory
Istnieje kilka dobrze znanych leków i toksyn, hamujących zachodzenie fosforylacji oksydacyjnej. Chociaż każda z tych substancji jest inhibitorem tylko jednego enzymu w łańcuchu transportu elektronów, inhibicja jednego z ogniw zatrzymuje cały proces. Na przykład oligomycyna jest inhibitorem syntazy ATP, powoduje, że protony nie mogą przemieścić się z powrotem do macierzy mitochondrialnej[83]. W efekcie kompleksy łańcucha oddechowego przenoszące protony przestają działać, ponieważ nie są w stanie pokonać wysokiego gradientu stężeń. NADH przestaje być utleniany, a cykl kwasu cytrynowego ustaje na skutek spadku stężenia NAD+ niezbędnego do działania części enzymów cyklu.
| Związek | Zastosowanie | Wpływ na fosforylację oksydacyjną |
|---|---|---|
| cyjanki, tlenek węgla | trucizny | Hamują zachodzenie łańcucha oddechowego poprzez zablokowanie centrum żelazo-miedź w oksydazie cytochromowej do którego przyłączany jest tlen. W efekcie elektrony nie są przenoszone na tlen[84]. |
| oligomycyna | antybiotyk | Jest inhibitorem kompleksu syntazy ATP, blokującym przenoszenie protonów przez domenę Fo[83]. |
| CCCP, 2,4-dinitrofenol | Jonofory przenoszące jony przez błonę lipidową, uniemożliwiając wytworzenie gradientu. W efekcie następuje rozprzężenie fosforylacji, ponieważ nie powstaje wystarczający do syntezy ATP gradient elektrochemiczny[85]. | |
| rotenon | pestycyd | Zatrzymuje przenoszenie elektronów z kompleksu I na ubichinon poprzez zablokowanie miejsca wiązania ubichononu[86]. |
| jabłczan i szczawiooctan | Są kompetytywnymi inhibitorami dehydrogenazy bursztynianowej (kompleksu II)[87]. |
Nie wszystkie inhibitory fosforylacji oksydacyjnej są toksynami. W brunatnej tkance tłuszczowej występują podlegające regulacji białka rozprzęgające (UCP), spełniające funkcję kanałów jonowych pozwalających na przejście przez błonę protonów, co prowadzi do rozprzężenia oddychania i syntezy ATP[88]. Szybkie zachodzenie oddychania, niepowiązane z fosforylacją, prowadzi do uwalniania energii w postaci ciepła, co pozwala utrzymać odpowiednią temperaturę ciała zwierzętom podczas snu zimowego. Białka rozprzęgające mogą także pełnić bardziej ogólną funkcję w komórkowej odpowiedzi na stres[89].
Historia badań
Badania nad fosforylacją oksydacyjną zapoczątkował Arthur Harden, który w roku 1906 stwierdził, że fosforan odgrywa ważną rolę w fermentacji przeprowadzanej przez komórki. Wcześniej wiedziano jedynie o włączaniu w proces fosforanów cukrów[90]. Na początku lat czterdziestych XX wieku zależność między utlenianiem węglowodanów a wytwarzaniem ATP została wykazana przez Hermana Kalckara[91], co potwierdzało tezę o kluczowej roli ATP w przenoszeniu energii, wysuniętej przez Fritza Lipmanna w roku 1941[92]. Następnie w roku 1949, Morris Friedkin i Albert L. Lehninger udowodnili, że koenzym NADH jest związkiem łączącym szlaki metaboliczne, cykl kwasu cytrynowego i syntezę ATP[93].
Przez kolejne dwadzieścia lat mechanizm wytwarzania ATP pozostawał tajemnicą, a badania naukowców koncertowały się na poszukiwaniu pośredniego związku bogatego w energię, który stanowiłby połączenie reakcji utleniania i fosforylacji[94]. Zagadka została rozwiązana przez Petera Mitchella publikacją teorii chemiosmotycznej w roku 1961[95]. Początkowo propozycja Mitchella wzbudzała wątpliwości, jednak doświadczalne potwierdzenie doprowadziło do akceptacji teorii i przyznanie jej twórcy nagrody Nobla w roku 1978[96][97]. Dalsze badania koncentrowały się na określeniu, wyizolowaniu i oczyszczeniu enzymów przeprowadzających poszczególne reakcji. Jednym z głównych badaczy zaangażowanych w poznawanie kompleksów łańcucha oddechowego był David E. Green, a kompleksu syntazy ATP Efraim Racker[98]. Przełomem w wyjaśnianiu mechanizmu syntezy ATP był zaproponowany przez Paula Boyera w roku 1973 mechanizm zmian konformacyjnych i jego rozwinięcie w postaci katalizowania syntezy ATP przez enzym w wyniku obracania się jego komponentów przedstawione w roku 1982[99][74]. W kolejnych latach badania nad strukturą enzymów uczestniczących w fosforylacji oksydacyjnej prowadził John E. Walker. Obaj, Walker i Boyer, za swoje prace otrzymali Nagrodę Nobla w roku 1997[100].
Zobacz też
- fosforylacja
- fosforylacja substratowa
- fosforylacja fotosyntetyczna
Przypisy
- ↑ Mitchell P, Moyle J. Chemiosmotic hypothesis of oxidative phosphorylation. „Nature”. 213 (5072), s. 137–139, 1967. DOI: 10.1038/213137a0. PMID: 4291593.
- ↑ Dimroth P, Kaim G, Matthey U. Crucial role of the membrane potential for ATP synthesis by F(1)F(o) ATP synthases. „J. Exp. Biol.”. 203 (Pt 1), s. 51–59, 2000. PMID: 10600673.
- ↑ a b c d Schultz B, Chan S. Structures and proton-pumping strategies of mitochondrial respiratory enzymes. „Annu Rev Biophys Biomol Struct”. 30, s. 23–65, 2001. DOI: 10.1146/annurev.biophys.30.1.23. PMID: 11340051.
- ↑ Rich PR. The molecular machinery of Keilin’s respiratory chain. „Biochem. Soc. Trans.”. 31 (Pt 6), s. 1095–1105, 2003. PMID: 14641005. [zarchiwizowane z adresu 2013-01-13].
- ↑ Porter RK, Brand MD. Mitochondrial proton conductance and H+/O ratio are independent of electron transport rate in isolated hepatocytes. „Biochem. J.”. 310 (Pt 2), s. 379–382, 1995. PMID: 7654171.
- ↑ Mathews FS. The structure, function and evolution of cytochromes. „Prog. Biophys. Mol. Biol.”. 45 (1), s. 1–56, 1985. DOI: 10.1016/0079-6107(85)90004-5. PMID: 3881803.
- ↑ Wood PM. Why do c-type cytochromes exist?. „FEBS Lett.”. 164 (2), s. 223–226, 1983. DOI: 10.1016/0014-5793(83)80289-0. PMID: 6317447.
- ↑ Crane FL. Biochemical functions of coenzyme Q10. „Journal of the American College of Nutrition”. 20 (6), s. 591–598, 2001. PMID: 11771674.
- ↑ Mitchell P. Keilin’s respiratory chain concept and its chemiosmotic consequences. „Science”. 206 (4423), s. 1148–1159, 1979. DOI: 10.1126/science.388618. PMID: 388618.
- ↑ Britta Søballe, Robert K. Poole, Microbial ubiquinones: multiple roles in respiration, gene regulation and oxidative stress management [pdf], „Microbiology (Reading, Engl.)”, 145 (Pt 8), 1999, s. 1817–1830, DOI: 10.1099/13500872-145-8-1817, PMID: 10463148 [dostęp 2009-02-18].
- ↑ Johnson D, Dean D, Smith A, Johnson M. Structure, function, and formation of biological iron-sulfur clusters. „Annu Rev Biochem”. 74, s. 247–281, 2005. DOI: 10.1146/annurev.biochem.74.082803.133518. PMID: 15952888.
- ↑ Page CC, Moser CC, Chen X, Dutton PL. Natural engineering principles of electron tunnelling in biological oxidation-reduction. „Nature”. 402 (6757), s. 47–52, 1999. DOI: 10.1038/46972. PMID: 10573417.
- ↑ Leys D, Scrutton NS. Electrical circuitry in biology: emerging principles from protein structure. „Curr. Opin. Struct. Biol.”. 14 (6), s. 642–647, 2004. DOI: 10.1016/j.sbi.2004.10.002. PMID: 15582386.
- ↑ Boxma B, de Graaf RM, van der Staay GW, et al. An anaerobic mitochondrion that produces hydrogen. „Nature”. 434 (7029), s. 74–79, 2005. DOI: 10.1038/nature03343. PMID: 15744302.
- ↑ van der Giezen M., Tovar J., Clark CG. Mitochondrion-derived organelles in protists and fungi. „International review of cytology”. 244, s. 175–225, 2005. DOI: 10.1016/S0074-7696(05)44005-X. PMID: 16157181.
- ↑ a b J. Hirst, Energy transduction by respiratory complex I–an evaluation of current knowledge, „Biochem. Soc. Trans.”, Pt 3, 33, 2005, s. 525–9, DOI: 10.1042/BST0330525, PMID: 15916556 [zarchiwizowane z adresu 2015-11-29].
- ↑ a b Lenaz G, Fato R, Genova M, Bergamini C, Bianchi C, Biondi A. Mitochondrial Complex I: structural and functional aspects. „Biochim Biophys Acta”. 1757 (9–10), s. 1406–1420, 2006. DOI: 10.1016/j.bbabio.2006.05.007. PMID: 16828051.
- ↑ a b Sazanov L.A., Hinchliffe P. (2006) Structure of the hydrophilic domain of respiratory complex I from Thermus thermophilus. Science 311, 1430–1436.
- ↑ Baranova EA, Holt PJ, Sazanov LA. Projection structure of the membrane domain of Escherichia coli respiratory complex I at 8 A resolution. „J. Mol. Biol.”. 366 (1), s. 140–154, 2007. DOI: 10.1016/j.jmb.2006.11.026. PMID: 17157874.
- ↑ Friedrich T, Böttcher B. The gross structure of the respiratory complex I: a Lego System. „Biochim. Biophys. Acta”. 1608 (1), s. 1–9, 2004. DOI: 10.1016/j.bbabio.2003.10.002. PMID: 14741580.
- ↑ Brandt U, Kerscher S, Dröse S, Zwicker K, Zickermann V. Proton pumping by NADH: ubiquinone oxidoreductase. A redox driven conformational change mechanism?. „FEBS Lett.”. 545 (1), s. 9–17, 2003. DOI: 10.1016/S0014-5793(03)00387-9. PMID: 12788486.
- ↑ Cecchini G. Function and structure of complex II of the respiratory chain. „Annu Rev Biochem”. 72, s. 77–109, 2003. DOI: 10.1146/annurev.biochem.72.121801.161700. PMID: 14527321.
- ↑ Yankovskaya V., Horsefield R., Tornroth S., Luna-Chavez C., Miyoshi H., Leger C., Byrne B., Cecchini G., Iwata S. (2003) Architecture of succinate dehydrogenase and reactive oxygen species generation. Science 299, 700–704.
- ↑ Horsefield R, Iwata S, Byrne B. Complex II from a structural perspective. „Curr. Protein Pept. Sci.”. 5 (2), s. 107–118, 2004. DOI: 10.2174/1389203043486847. PMID: 15078221.
- ↑ Kita K, Hirawake H, Miyadera H, Amino H, Takeo S. Role of complex II in anaerobic respiration of the parasite mitochondria from Ascaris suum and Plasmodium falciparum. „Biochim. Biophys. Acta”. 1553 (1–2), s. 123–139, 2002. DOI: 10.1016/S0005-2728(01)00237-7. PMID: 11803022.
- ↑ Painter HJ, Morrisey JM, Mather MW, Vaidya AB. Specific role of mitochondrial electron transport in blood-stage Plasmodium falciparum. „Nature”. 446 (7131), s. 88–91, 2007. DOI: 10.1038/nature05572. PMID: 17330044.
- ↑ Ramsay RR, Steenkamp DJ, Husain M. Reactions of electron-transfer flavoprotein and electron-transfer flavoprotein: ubiquinone oxidoreductase. „Biochem. J.”. 241 (3), s. 883–892, 1987. PMID: 3593226.
- ↑ Zhang J, Frerman FE, Kim JJ. Structure of electron transfer flavoprotein-ubiquinone oxidoreductase and electron transfer to the mitochondrial ubiquinone pool. „Proc. Natl. Acad. Sci. U.S.A.”. 103 (44), s. 16212–16217, 2006. DOI: 10.1073/pnas.0604567103. PMID: 17050691.
- ↑ Ikeda Y, Dabrowski C, Tanaka K. Separation and properties of five distinct acyl-CoA dehydrogenases from rat liver mitochondria. Identification of a new 2-methyl branched chain acyl-CoA dehydrogenase. „J. Biol. Chem.”. 258 (2), s. 1066–1076, 1983. PMID: 6401712.
- ↑ F.J. Ruzicka, H. Beinert, A new iron-sulfur flavoprotein of the respiratory chain. A component of the fatty acid beta oxidation pathway [pdf], „Journal of Biological Chemistry”, 252 (23), 1977, s. 8440–5, PMID: 925004.
- ↑ Ishizaki K, Larson TR, Schauer N, Fernie AR, Graham IA, Leaver CJ. The critical role of Arabidopsis electron-transfer flavoprotein:ubiquinone oxidoreductase during dark-induced starvation. „Plant Cell”. 17 (9), s. 2587–2600, 2005. DOI: 10.1105/tpc.105.035162. PMID: 16055629.
- ↑ Berry E, Guergova-Kuras M, Huang L, Crofts A. Structure and function of cytochrome bc complexes. „Annu Rev Biochem”. 69, s. 1005–1075, 2000. DOI: 10.1146/annurev.biochem.69.1.1005. PMID: 10966481.
- ↑ Crofts AR. The cytochrome bc1 complex: function in the context of structure. „Annu. Rev. Physiol.”. 66, s. 689–733, 2004. DOI: 10.1146/annurev.physiol.66.032102.150251. PMID: 14977419.
- ↑ Iwata S, Lee JW, Okada K, et al. Complete structure of the 11-subunit bovine mitochondrial cytochrome bc1 complex. „Science”. 281 (5373), s. 64–71, 1998. DOI: 10.1126/science.281.5373.64. PMID: 9651245.
- ↑ B.L. Trumpower, The protonmotive Q cycle. Energy transduction by coupling of proton translocation to electron transfer by the cytochrome bc1 complex [pdf], „Journal of Biological Chemistry”, 265 (20), 1990, s. 11409–12, PMID: 2164001.
- ↑ Hunte C, Palsdottir H, Trumpower BL. Protonmotive pathways and mechanisms in the cytochrome bc1 complex. „FEBS Lett.”. 545 (1), s. 39–46, 2003. DOI: 10.1016/S0014-5793(03)00391-0. PMID: 12788490.
- ↑ Calhoun M, Thomas J, Gennis R. The cytochrome oxidase superfamily of redox-driven proton pumps. „Trends Biochem Sci”. 19 (8), s. 325–330, 1994. DOI: 10.1016/0968-0004(94)90071-X. PMID: 7940677.
- ↑ Tsukihara T, Aoyama H, Yamashita E, Tomizaki T, Yamaguchi H, Shinzawa-Itoh K, Nakashima R, Yaono R, Yoshikawa S. TThe whole structure of the 13-subunit oxidized cytochrome c oxidase at 2.8 A. „Science”. 272 (5265), s. 1136–1144, 1996. DOI: 10.1126/science.272.5265.1136. PMID: 8638158.
- ↑ Yoshikawa S, Muramoto K, Shinzawa-Itoh K, et al. Proton pumping mechanism of bovine heart cytochrome c oxidase. „Biochim. Biophys. Acta”. 1757 (9–10), s. 1110–1116, 2006. DOI: 10.1016/j.bbabio.2006.06.004. PMID: 16904626.
- ↑ Rasmusson AG, Soole KL, Elthon TE. Alternative NAD(P)H dehydrogenases of plant mitochondria. „Annual review of plant biology”. 55, s. 23–39, 2004. DOI: 10.1146/annurev.arplant.55.031903.141720. PMID: 15725055.
- ↑ Menz RI, Day DA. Purification and characterization of a 43-kDa rotenone-insensitive NADH dehydrogenase from plant mitochondria. „J. Biol. Chem.”. 271 (38), s. 23117–23120, 1996. DOI: 10.1074/jbc.271.38.23117. PMID: 8798503.
- ↑ McDonald A, Vanlerberghe G. Branched mitochondrial electron transport in the Animalia: presence of alternative oxidase in several animal phyla. „IUBMB Life”. 56 (6), s. 333–341, 2004. DOI: 10.1080/1521-6540400000876. PMID: 15370881.
- ↑ Sluse FE, Jarmuszkiewicz W. Alternative oxidase in the branched mitochondrial respiratory network: an overview on structure, function, regulation, and role. „Braz. J. Med. Biol. Res.”. 31 (6), s. 733–747, 1998. DOI: 10.1590/S0100-879X1998000600003. PMID: 9698817.
- ↑ Moore AL, Siedow JN. The regulation and nature of the cyanide-resistant alternative oxidase of plant mitochondria. „Biochim. Biophys. Acta”. 1059 (2), s. 121–140, 1991. DOI: 10.1016/S0005-2728(05)80197-5. PMID: 1883834.
- ↑ Vanlerberghe GC, McIntosh L. Alternative oxidase: From Gene to Function. „Annual Review of Plant Physiology and Plant Molecular Biology”. 48, s. 703–734, 1997. DOI: 10.1146/annurev.arplant.48.1.703. PMID: 15012279.
- ↑ Ito Y, Saisho D, Nakazono M, Tsutsumi N, Hirai A. Transcript levels of tandem-arranged alternative oxidase genes in rice are increased by low temperature. „Gene”. 203 (2), s. 121–129, 1997. DOI: 10.1016/S0378-1119(97)00502-7. PMID: 9426242.
- ↑ Maxwell DP, Wang Y, McIntosh L. The alternative oxidase lowers mitochondrial reactive oxygen production in plant cells. „Proc. Natl. Acad. Sci. U.S.A.”. 96 (14), s. 8271–8276, 1999. DOI: 10.1073/pnas.96.14.8271. PMID: 10393984.
- ↑ Lenaz G. A critical appraisal of the mitochondrial coenzyme Q pool. „FEBS Lett.”. 509 (2), s. 151–155, 2001. DOI: 10.1016/S0014-5793(01)03172-6. PMID: 11741580.
- ↑ Heinemeyer J, Braun HP, Boekema EJ, Kouril R. A structural model of the cytochrome C reductase/oxidase supercomplex from yeast mitochondria. „J. Biol. Chem.”. 282 (16), s. 12240–12248, 2007. DOI: 10.1074/jbc.M610545200. PMID: 17322303.
- ↑ Schägger H, Pfeiffer K. Supercomplexes in the respiratory chains of yeast and mammalian mitochondria. „EMBO J.”. 19 (8), s. 1777–1783, 2000. DOI: 10.1093/emboj/19.8.1777. PMID: 10775262.
- ↑ Schägger H. Respiratory chain supercomplexes of mitochondria and bacteria. „Biochim. Biophys. Acta”. 1555 (1–3), s. 154–159, 2002. DOI: 10.1016/S0005-2728(02)00271-2. PMID: 12206908.
- ↑ Schägger H, Pfeiffer K. The ratio of oxidative phosphorylation complexes I-V in bovine heart mitochondria and the composition of respiratory chain supercomplexes. „J. Biol. Chem.”. 276 (41), s. 37861–37867, 2001. DOI: 10.1074/jbc.M106474200. PMID: 11483615.
- ↑ Gupte S, Wu ES, Hoechli L, et al. Relationship between lateral diffusion, collision frequency, and electron transfer of mitochondrial inner membrane oxidation-reduction components. „Proc. Natl. Acad. Sci. U.S.A.”. 81 (9), s. 2606–2610, 1984. DOI: 10.1073/pnas.81.9.2606. PMID: 6326133.
- ↑ Nealson KH. Post-Viking microbiology: new approaches, new data, new insights. „Origins of life and evolution of the biosphere: the journal of the International Society for the Study of the Origin of Life”. 29 (1), s. 73–93, 1999. DOI: 10.1023/A:1006515817767. PMID: 11536899.
- ↑ Schäfer G, Engelhard M, Müller V. Bioenergetics of the Archaea. „Microbiol. Mol. Biol. Rev.”. 63 (3), s. 570–620, 1999. PMID: 10477309.
- ↑ a b Ingledew WJ, Poole RK. The respiratory chains of Escherichia coli. „Microbiol. Rev.”. 48 (3), s. 222–271, 1984. PMID: 6387427.
- ↑ a b Unden G, Bongaerts J. Alternative respiratory pathways of Escherichia coli: energetics and transcriptional regulation in response to electron acceptors. „Biochim. Biophys. Acta”. 1320 (3), s. 217–234, 1997. DOI: 10.1016/S0005-2728(97)00034-0. PMID: 9230919.
- ↑ Cecchini G, Schröder I, Gunsalus RP, Maklashina E. Succinate dehydrogenase and fumarate reductase from Escherichia coli. „Biochim. Biophys. Acta”. 1553 (1–2), s. 140–157, 2002. DOI: 10.1016/S0005-2728(01)00238-9. PMID: 11803023.
- ↑ Freitag A, Bock E. Energy conservation in Nitrobacter. „FEMS Microbiology Letters”. 66 (1–3), s. 157–162, 1990. DOI: 10.1111/j.1574-6968.1990.tb03989.x.
- ↑ Starkenburg SR, Chain PS, Sayavedra-Soto LA, et al. Genome sequence of the chemolithoautotrophic nitrite-oxidizing bacterium Nitrobacter winogradskyi Nb-255. „Appl. Environ. Microbiol.”. 72 (3), s. 2050–2063, 2006. DOI: 10.1128/AEM.72.3.2050-2063.2006. PMID: 16517654.
- ↑ Yamanaka T, Fukumori Y. The nitrite oxidizing system of Nitrobacter winogradskyi. „FEMS Microbiol. Rev.”. 4 (4), s. 259–270, 1988. PMID: 2856189.
- ↑ Iuchi S, Lin EC. Adaptation of Escherichia coli to redox environments by gene expression. „Mol. Microbiol.”. 9 (1), s. 9–15, 1993. DOI: 10.1111/j.1365-2958.1993.tb01664.x. PMID: 8412675.
- ↑ M.W. Calhoun i inni, Energetic efficiency of Escherichia coli: effects of mutations in components of the aerobic respiratory chain [pdf], „Journal of Bacteriology”, 175 (10), 1993, s. 3020–5, PMID: 8491720.
- ↑ a b Boyer PD. The ATP synthase–a splendid molecular machine. „Annu. Rev. Biochem.”. 66, s. 717–749, 1997. DOI: 10.1146/annurev.biochem.66.1.717. PMID: 9242922.
- ↑ Van Walraven HS, Strotmann H, Schwarz O, Rumberg B. The H+/ATP coupling ratio of the ATP synthase from thiol-modulated chloroplasts and two cyanobacterial strains is four. „FEBS Lett.”. 379 (3), s. 309–313, 1996. DOI: 10.1016/0014-5793(95)01536-1. PMID: 8603713.
- ↑ Yoshida M, Muneyuki E, Hisabori T. ATP synthase–a marvellous rotary engine of the cell. „Nat. Rev. Mol. Cell Biol.”. 2 (9), s. 669–677, 2001. DOI: 10.1038/35089509. PMID: 11533724.
- ↑ Schemidt RA, Qu J, Williams JR, Brusilow WS. Effects of carbon source on expression of F0 genes and on the stoichiometry of the c subunit in the F1F0 ATPase of Escherichia coli. „J. Bacteriol.”. 180 (12), s. 3205–3208, 1998. PMID: 9620972.
- ↑ Nelson N, Perzov N, Cohen A, Hagai K, Padler V, Nelson H. The cellular biology of proton-motive force generation by V-ATPases. „J. Exp. Biol.”. 203 (Pt 1), s. 89–95, 2000. PMID: 10600677.
- ↑ Rubinstein JL, Walker JE, Henderson R. Structure of the mitochondrial ATP synthase by electron cryomicroscopy. „EMBO J.”. 22 (23), s. 6182–6192, 2003. DOI: 10.1093/emboj/cdg608. PMID: 14633978.
- ↑ Leslie AG, Walker JE. Structural model of F1-ATPase and the implications for rotary catalysis. „Philos. Trans. R. Soc. Lond., B, Biol. Sci.”. 355 (1396), s. 465–471, 2000. DOI: 10.1098/rstb.2000.0588. PMID: 10836500.
- ↑ Noji H, Yoshida M. The rotary machine in the cell, ATP synthase. „J. Biol. Chem.”. 276 (3), s. 1665–1668, 2001. DOI: 10.1074/jbc.R000021200. PMID: 11080505.
- ↑ Capaldi R, Aggeler R. Mechanism of the F(1)F(0)-type ATP synthase, a biological rotary motor. „Trends Biochem Sci”. 27 (3), s. 154–160, 2002. DOI: 10.1016/S0968-0004(01)02051-5. PMID: 11893513.
- ↑ Dimroth P, von Ballmoos C, Meier T. Catalytic and mechanical cycles in F-ATP synthases. Fourth in the Cycles Review Series. „EMBO Rep”. 7 (3), s. 276–282, 2006. DOI: 10.1038/sj.embor.7400646. PMID: 16607397.
- ↑ a b Gresser MJ, Myers JA, Boyer PD. Catalytic site cooperativity of beef heart mitochondrial F1 adenosine triphosphatase. Correlations of initial velocity, bound intermediate, and oxygen exchange measurements with an alternating three-site model. „J. Biol. Chem.”. 257 (20), s. 12030–12038, 1982. PMID: 6214554.
- ↑ Dimroth P. Bacterial sodium ion-coupled energetics. „Antonie Van Leeuwenhoek”. 65 (4), s. 381–395, 1994. DOI: 10.1007/BF00872221. PMID: 7832594.
- ↑ a b Becher B, Müller V. Delta mu Na+ drives the synthesis of ATP via a delta mu Na(+)-translocating F1F0-ATP synthase in membrane vesicles of the archaeon Methanosarcina mazei Gö1. „J. Bacteriol.”. 176 (9), s. 2543–2550, 1994. PMID: 8169202.
- ↑ Müller V. An exceptional variability in the motor of archaeal A1A0 ATPases: from multimeric to monomeric rotors comprising 6–13 ion binding sites. „J. Bioenerg. Biomembr.”. 36 (1), s. 115–125, 2004. DOI: 10.1023/B:JOBB.0000019603.68282.04. PMID: 15168615.
- ↑ a b Davies K. Oxidative stress: the paradox of aerobic life. „Biochem Soc Symp”. 61, s. 1–31, 1995. PMID: 8660387.
- ↑ Rattan SI. Theories of biological aging: genes, proteins, and free radicals. „Free Radic. Res.”. 40 (12), s. 1230–1238, 2006. DOI: 10.1080/10715760600911303. PMID: 17090411.
- ↑ Valko M, Leibfritz D, Moncol J, Cronin MT, Mazur M, Telser J. Free radicals and antioxidants in normal physiological functions and human disease. „Int. J. Biochem. Cell Biol.”. 39 (1), s. 44–84, 2007. DOI: 10.1016/j.biocel.2006.07.001. PMID: 16978905.
- ↑ Raha S, Robinson B. Mitochondria, oxygen free radicals, disease and ageing. „Trends Biochem Sci”. 25 (10), s. 502–508, 2000. DOI: 10.1016/S0968-0004(00)01674-1. PMID: 11050436.
- ↑ Finkel T, Holbrook NJ. Oxidants, oxidative stress and the biology of ageing. „Nature”, s. 239–247, 2000. DOI: 10.1038/35041687. PMID: 11089981.
- ↑ a b Joshi S, Huang YG. ATP synthase complex from bovine heart mitochondria: the oligomycin sensitivity conferring protein is essential for dicyclohexyl carbodiimide-sensitive ATPase. „Biochim. Biophys. Acta”. 1067 (2), s. 255–258, 1991. DOI: 10.1016/0005-2736(91)90051-9. PMID: 1831660.
- ↑ Tsubaki M. Fourier-transform infrared study of cyanide binding to the Fea3-CuB binuclear site of bovine heart cytochrome c oxidase: implication of the redox-linked conformational change at the binuclear site. „Biochemistry”. 32 (1), s. 164–173, 1993. DOI: 10.1021/bi00052a022. PMID: 8380331.
- ↑ Heytler PG. Uncouplers of oxidative phosphorylation. „Meth. Enzymol.”. 55, s. 462–442, 1979. DOI: 10.1016/0076-6879(79)55060-5. PMID: 156853.
- ↑ Lambert AJ, Brand MD. Inhibitors of the quinone-binding site allow rapid superoxide production from mitochondrial NADH: ubiquinone oxidoreductase (complex I). „J. Biol. Chem.”. 279 (38), s. 39414–39420, 2004. DOI: 10.1074/jbc.M406576200. PMID: 15262965.
- ↑ Dervartanian DV, Veeger C. Studies on succinate dehydrogenase. I. Spectral properties of the purified enzyme and formation of enzyme-competitive inhibitor complexes. „Biochim. Biophys. Acta”. 92, s. 233–247, November 1964. PMID: 14249115.
- ↑ Ricquier D, Bouillaud F. The uncoupling protein homologues: UCP1, UCP2, UCP3, StUCP and AtUCP. „Biochem. J.”. 345 Pt 2, s. 161–179, 2000. DOI: 10.1042/0264-6021:3450161. PMID: 10620491.
- ↑ Borecký J, Vercesi AE. Plant uncoupling mitochondrial protein and alternative oxidase: energy metabolism and stress. „Biosci. Rep.”. 25 (3–4), s. 271–286, 2005. DOI: 10.1007/s10540-005-2889-2. PMID: 16283557.
- ↑ Harden A, Young WJ. The alcoholic ferment of yeast-juice. „Proc. R. Soc. (Lond.)”. B (77), s. 405–420, 1906.
- ↑ Kalckar HM. Origins of the concept oxidative phosphorylation. „Mol. Cell. Biochem.”. 5 (1–2), s. 55–63, 1974. DOI: 10.1007/BF01874172. PMID: 4279328.
- ↑ Lipmann F. Metabolic generation and utilization of phosphate bond energy. „Adv Enzymol”. 1, s. 99–162, 1941.
- ↑ Friedkin M, Lehninger AL. Esterification of inorganic phosphate coupled to electron transport between dihydrodiphosphopyridine nucleotide and oxygen. „J. Biol. Chem.”. 178 (2), s. 611–623, 1949.
- ↑ Slater EC. Mechanism of Phosphorylation in the Respiratory Chain. „Nature”. 172 (4387), s. 975, 1953. DOI: 10.1038/172975a0.
- ↑ Mitchell P. Coupling of Phosphorylation to Electron and Hydrogen Transfer by a Chemi-Osmotic type of Mechanism. „Nature”. 191 (4784), s. 144, 1961. DOI: 10.1038/191144a0. PMID: 13771349.
- ↑ Milton H. Saier Jr: Peter Mitchell and the Vital Force. [dostęp 2007-08-23].
- ↑ Peter Mitchell: David Keilin’s Respiratory Chain Concept and Its Chemiosmotic Consequences. [w:] Nobel lecture [on-line]. Nobel Foundation, 1978. [dostęp 2007-07-21].
- ↑ Pullman ME, Penefsky HS, Datta A, and Racker E. Partial Resolution of the Enzymes Catalyzing Oxidative Phosphorylation. I. Purification and Properties of Soluble, Dinitrophenol-stimulated Adenosine Triphosphatase. „J. Biol. Chem.”. 235 (11), s. 3322–3329, 1960.
- ↑ Boyer PD, Cross RL, Momsen W. A new concept for energy coupling in oxidative phosphorylation based on a molecular explanation of the oxygen exchange reactions. „Proc. Natl. Acad. Sci. U.S.A.”. 70 (10), s. 2837–2839, 1973. DOI: 10.1073/pnas.70.10.2837. PMID: 4517936.
- ↑ The Nobel Prize in Chemistry 1997. Nobel Foundation. [dostęp 2007-07-21].
Bibliografia
- Jeremy Mark Berg, John L Tymoczko, Lubert Stryer, Neil D Clarke, Zofia Szweykowska-Kulińska, Artur Jarmołowski, Halina Augustyniak: Biochemia. Warszawa: Wydawnictwo Naukowe PWN, 2007. ISBN 978-83-01-14379-4.
Linki zewnętrzne
- Fosforylacja oksydacyjna. www2.ufp.pt. [zarchiwizowane z tego adresu (2014-09-02)]. (ang.)
Media użyte na tej stronie
Schematic diagram of electron transport in complex IV (cytochrome c oxidase) of the mitochondrial electron transport chain.
Schematic diagram of electron transport in complex I (NADH dehydrogenase) of the mitochondrial electron transport chain.
Conversion of ubiquinone to ubiquinol. Created using ACD/ChemSketch 10.0 and Inkscape.
Schematic diagram of electron transport in complex II (succinate–coenzyme Q reductase, succinate dehydrogenase) of the mitochondrial electron transport chain.
Schematic diagram of the mitochondrial electron transport chain.
Complexo III da cadeia de transporte electrónico na fosforilação oxidativa.