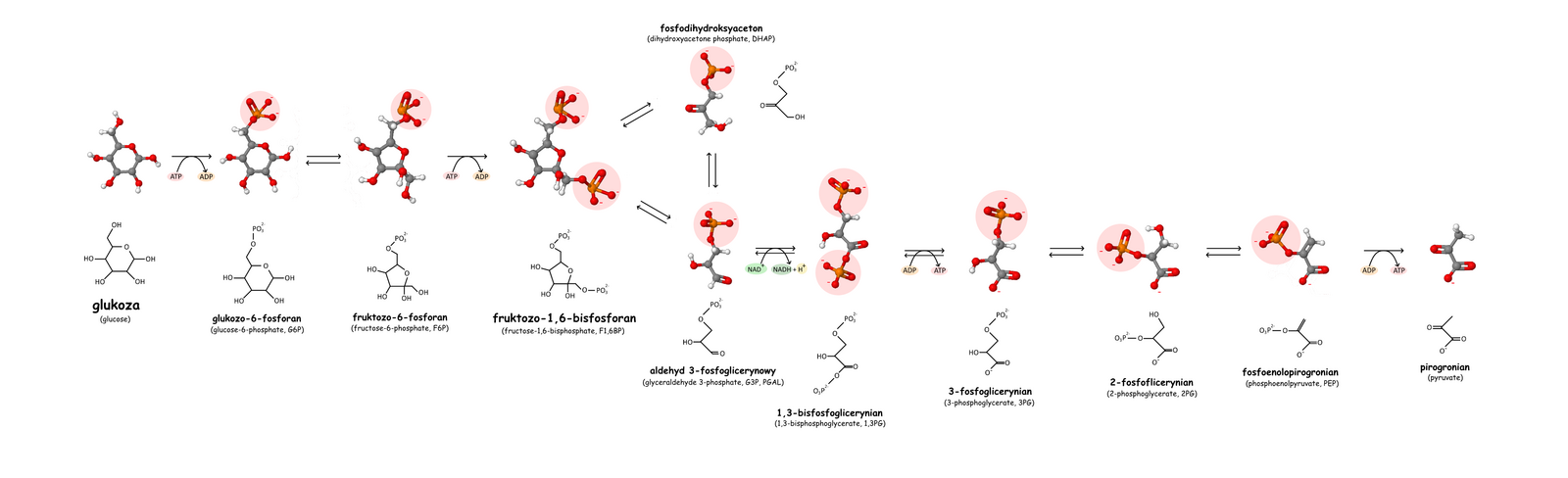
Glikoliza
Glikoliza, szlak Embdena-Meyerhofa-Parnasa – ciąg reakcji biochemicznych, podczas których jedna cząsteczka glukozy zostaje przekształcona w dwie cząsteczki pirogronianu.
Glikoliza jest ewolucyjnie starym szlakiem metabolicznym, szeroko rozpowszechnionym wśród organizmów żywych należących do wszystkich trzech domen[1][2]. U eukariotów i wielu prokariotów; przebiega w cytoplazmie podstawowej[3] oraz w plastydach obecnych w komórkach roślin[4][5]. U niektórych protistów[6], takich jak Trypanosoma[7][8] i Leishmania[9] glikoliza zachodzi w wyspecjalizowanych organellach nazywanych glikosomami[10][11].
Najczęstszym typem glikolizy jest schemat Embden-Meyerhof-Parnas, który został odkryty przez Gustava Embdena, Otto Meyerhofa i Jakuba Karola Parnasa.
Sumaryczna reakcja glikolizy jest następująca:
- glukoza + 2 Pi + 2 ADP + 2 NAD+ → 2 cząsteczki pirogronianu + 2 ATP + 2 NADH + 2 H+ + 2 H2O
Rola
Rolą glikolizy jest:
- dostarczanie energii – w wyniku glikolizy powstają 2 cząsteczki ATP oraz substraty do cyklu kwasu cytrynowego i fosforylacji oksydacyjnej, gdzie wytwarzana jest większa ilość ATP.
- wytwarzanie intermediatów dla szlaków biosyntetycznych.
Etapy
Fosforylacja glukozy
 | ATP ADP Heksokinaza lub Glukokinaza | 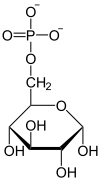 |
| α-D-Glukoza | α-D-Glukozo-6-fosforan |
Fosforylacja glukozy i powstanie glukozo-6-fosforanu jest reakcją nieodwracalną katalizowaną przez heksokinazę lub bardziej specyficznie w wątrobie przez glukokinazę[13]. Jako dawca fosforanu potrzebny jest do tej reakcji ATP, reagujący w formie kompleksu Mg-ATP. Jon magnezu Mg2+ lub dwuwartościowy jon innego metalu, na przykład Mn2+, jest konieczny do aktywności heksokinazy. Enzym ten składa się z dwóch części, które podczas połączenia z glukozą zbliżają się do siebie. W wyniku zbliżenia cząsteczka glukozy zostaje zamknięta wewnątrz białka. Indukowane przez glukozę zmiany struktury enzymu powodują, że środowisko wokół cząsteczki cukru staje się mniej polarne. Jedynym elementem wystającym na zewnątrz enzymu jest węgiel C-6 glukozy z grupą hydroksylową. Obniżenie polarności ułatwia przeniesienie grupy fosforanowej z ATP na węgiel C-6. Zamykanie szczeliny indukowane substratem zapobiega reakcji hydrolizy ATP przy użyciu H2O. Reakcja taka zachodziłaby, gdyby miejsce atomu C-6 z grupą hydroksylową mogło być zajęte przez cząsteczkę wody[3].
W komórkach roślinnych występuje wiele rodzajów heksokinaz. Większość z nich to enzymy cytozolowe zdolne do fosforylacji zarówno glukozy, jak i innych heksoz w tym fruktozy i mannozy. W niektórych tkankach wstępujące heksokinaza związana zewnętrzną błoną mitochondrialną, fosforylująca przede wszystkim glukozę[14].
Izomeryzacja
 | Izomeraza glukozo- fosforanowa  |  |
| α-D-Glukozo-6-fosforan | α-D-Fruktozo-6-fosforan |
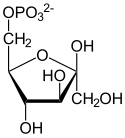
Przekształcenie glukozo-6-fosforanu we fruktozo-6-fosforan następuje przy udziale izomerazy glukozo-6-fosforanowej[13], z zastrzeżeniem, że przemianie tej ulega tylko anomer α glukozo-6-fosforanu. Reakcja polega na przekształceniu aldozy, którą jest glukoza z grupą aldehydową przy C-1, do ketozy, którą jest fruktoza z grupą ketonową przy C-2. Pierwszy etap reakcji polega na otworzeniu formy pierścieniowej glukozo-6-fosforanu. Dopiero otwarta forma łańcuchowa ulega izomeryzacji do formy łańcuchowej fruktozo-6-fosforanu. Ostatni etap to przekształcenie otwartej formy łańcuchowej do pięcioczłonowej formy pierścieniowej[3].
Fosforylacja fruktozo-6-fosforanu
 | ATP ADP Fosfo- frukto- kinaza | 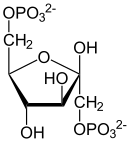 |
| β-D-Fruktozo-6-fosforan | β-D-Fruktozo-1,6-bisfosforan |
Fosforylację fruktozo-6-fosforanu przy użyciu ATP przeprowadza enzym fosfofruktokinaza I (PFK-1, ATP-fosfofruktokinaza), powstaje fruktozo-1,6-bisfosforanu oraz ADP[13]. Reakcja ta jest nieodwracalna w warunkach fizjologicznych.
W komórkach roślin wyższych i części niższych fosforylacja fruktozo-6-fosforanu może zachodzić przy udziale fosfofruktokinazy lub 1-fosfotransferazy pirofosforan-fruktozo-6-fosforan (PPi-fosfofruktokinaza). Ten drugi enzym przeprowadza reakcję fosforylacji zużywając nie ATP, lecz pirofosforan, a katalizowana reakcja jest odwracalna. W tkankach młodych fruktozo-1,6-bisfosforan powstaje głównie w wyniku reakcji przeprowadzanej przez PPi-fosfofruktokinazę, a w tkankach starszych ATP-fosfofruktokinazę[14].
Rozszczepienie fruktozo-1,6-bisfosforanu

 | Aldolaza |  |  | 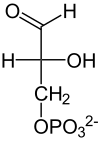 |
| β-D-Fruktozo-1,6-bisfosforan | Fosfo- dihydroksy- aceton | D-Aldehyd- 3-fosfo- glicerynowy |
Rozszczepienie fruktozo-1,6-bisfosforanu na dwie fosfotriozy – aldehyd 3-fosfoglicerynowy oraz fosfodihydroksyaceton przeprowadzane jest przez aldolazę[13]. Enzym ten katalizuje reakcję kondensacji aldolowej oraz reakcję odwrotną. Nazwa pochodzi od reakcji kondensacji, jednak w glikolizie dochodzi do rozszczepienia sześciowęglowego cukru[3]. Znane są dwie klasy aldolazy fruktozo-1,6-bisfosforanu (EC 4.1.2.13) o różnym mechanizmie enzymatycznym. Klasa I przeprowadza reakcję poprzez wytworzenie zasady Schiffa pomiędzy grupą karbonylową substratów a grupą aminowa lizyny znajdującej się w centrum aktywnym enzymu. Klasa druga stabilizuje związek pośredni w postaci karboanionu za pomocą dwuwartościowego jonu metalu. Enzymy należące do klasy I nie są jednorodne. U eukariontów to najczęściej homotetramer o masie podjednostki około 40 kDa, a u bakterii są zróżnicowanymi oligomerami o masie podjednostek pomiędzy 27-4- kDa. Aldolazy należące do klasy II występujące u bakterii i eukariontów zwykle są dimerami o masie podjednostek około 40 kDa[16]. U archeanów występują aldolazy zaliczane do klasy I oraz klasy II[17][16]. Różnią się one jednak znacznie od enzymów występujących w pozostałych domenach[16]. W plastydach najczęściej występuje aldolaza klasy I, jednak u niektórych organizmów stwierdzono występowanie w plastydach aldolazy klasy II[18]. Z badań nad aldolazą klasy II u Escheriachia coli wynika, że kluczowe dla funkcji enzymu są reszty aminokwasów Asn-35, Ser,-61 i Lys-325. Ser-61 bierze udział w wiązaniu aldehydu 3-fosfoglicerynowego. Pozostałe dwie reszty mają wpływ na zdolności katalityczne enzymu[19].
Dziedziczny niedobór aldolazy w erytrocytach może wywoływać niedokrwistość hemolityczną.
Przekształcenie fosfodihydroksyacetonu w aldehyd 3-fosfoglicerynowy
 | Izomeraza triozo- fosforanowa  |  |
| Fosfo- dihydroksy- aceton | D-Aldehyd- 3-fosfo- glicerynowy |
Przekształcenie fosfodihydroksyacetonu w aldehyd 3-fosfoglicerynowy przez izomerazę triozofosforanową[13]. W stanie równowagi 96% triozofosforanów występuje w postaci fosfodihydroksyacetonu. Mimo to dzięki izomerazie triozofosforanowej (TIM) możliwe jest szybkie wytworzenie aldehydu 3-fosfoglicerynowego niezbędnego do dalszego zachodzenia glikolizy. Katalizowana wewnątrzcząsteczkowa reakcja oksydoredukcyjna polega na przeniesieniu atomu wodoru z C-1 na C-2. W oderwaniu protonu od atomu węgla bierze udział Glu-165, zaś His-95 dostarcza proton grupie karbonylowej C-2 stabilizując jej ładunek ujemny. Produktem pośrednim reakcji jest endiol. Enzym zapobiega rozpadowi endiolowego związku pośredniego na metyloglioksal i ortofosforan, nie pozwalając związkowi endiolowemu na oderwanie się. W utrzymywaniu produktu pośredniego uczestniczy 10 reszt aminokwasowych TIM, zamykających endiol w centrum aktywnym[3].
Utlenianie i fosforylacja aldehydu 3-fosfoglicerynowego
 | NAD+ NADH + Pi + H+  Dehydrogenaza aldehydu- 3-fosfoglicerynowego | 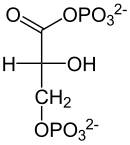 |
| D-Aldehyd 3-fosfoglicerynowy | D-1,3-bisfosfoglicerynian |
Przekształcenie aldehydu 3-fosfoglicerynowego w 1,3-bisfosfoglicerynian (1,3-BPG) z użyciem fosforanu nieorganicznego, NAD+ i enzymu dehydrogenazy aldehydu 3-fosfoglicerynowego[13]. Jest to jednoczesna reakcja utleniania i fosforylacji, która może być zmodyfikowana w obecności arsenianu – reaguje on z nieorganicznym fosforanem i tworzy 1-arseno-3-fosfoglicerynian i – zamiast ATP – energię cieplną. Reakcja utleniania jest korzystna termodynamicznie i wykazuje zmianę standardowej energii swobodnej ΔG°' około -50 kJ mol−1. Reakcja ta przebiega w dwóch etapach. W pierwszym z nich aldehyd 3-fosfoglicerynowy łącząć się z grupą tiolową Cys-149 tworzy hemitioacetal. W drugim etapie jon wodorkowy przenoszony jest na NAD+ połączonym z enzymem w pobliżu reszty cysteiny. W oderwaniu protonu od hemiacetalu bierze udział His-176. Druga reakcja wiąże się ze zmianą standardowej energii swobodnej o podobną wartość z przeciwnym znakiem. Sprzężenie obu reakcji jest możliwe dzięki produktowi pośredniemu o charakterze tioestru. Tioestrowy związek pośredni reaguje z ortofosforanem tworząc acylofosforan. W wyniku połączenia z ortofosforanem zostaje uwolniona reszta Cys-149 i powstaje 1,3-bisfosfoglicerynian. Do połączenia może jednak dość tylko gdy powstały w pierwszej reakcji NADH odłączy się od enzymu, a w jego miejsce przyłączy się kolejna cząsteczka NAD+. To właśnie dodatni ładunek dinukleotydu nikotynoamidoadeninowego ułatwia przyłączenie ortofosforanu. Energetyczne sprzężenie obu reakcji jest kluczowe dla pozyskania energii użytecznej metabolicznie w postaci ATP[3].
Synteza ATP 1
 | Kinaza fosfo- glicerynianowa ADP ATP  | 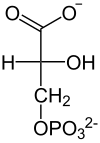 |
| D-1,3-bisfosfoglicerynian | D-3-fosfoglicerynian |
Przeniesienie grupy fosforanowej z 1,3-BPG na ADP i utworzenie ATP (fosforylacja substratowa) oraz 3-fosfoglicerynianu – reakcja katalizowana przez kinazę fosfoglicerynianową[21]. Enzym jest monomerem o masie 45 kDa i wysokim stopniu podobieństwa u różnych gatunków. Enzymy z mięśni konia i drożdży są niemal identyczne[22]. Podobnie jak u innych kinaz substraty przyłączane są do dwóch domen. Pierwsza łączy się z 1,3-bisfosfoglicerynianem a druga z kompleksem Mg-ADP[23][24]. W wyniku zmian konformacyjnych dochodzi do zbliżenia obu substratów i przeniesienia grupy fosforanowej z pozycji C-1 1,3-bisfosfoglicerynianu na ADP[25][26] Reakcja jest odwracalna[25].
Przesunięcie fosforanu
 | Fosfo- glicero- mutaza  |  |
| D-3-fosfoglicerynian | D-2-fosfoglicerynian |
Przekształcenie 3-fosfoglicerynianu w 2-fosfoglicerynian przez fosfogliceromutazę[13]. Przesunięcie grupy fosforanowej odbywa się poprzez przyłączenie do 3-fosfoglicerynianu fosforanu pochodzącego z reszty histydyny enzymu. W efekcie powstaje 2,3-bisfosfoglicerynian (2,3-BPG). Związek ten służy następnie do ufosforylowania reszty histydyny. Na enzym przenoszona jest grupa z pozycji 3. Do działania enzymu potrzebna jest katalityczna ilość 2,3-bisfosfoglicerynianu[3].
Odwodnienie
 | –H2O Enolaza  |  |
| D-2-fosfoglicerynian | Fosfoenolopirogronian |
Odwodnienie 2-fosfoglicerynianu i powstanie fosfoenolopirogronianu (PEP) katalizowane jest przez enolazę (EC 4.2.1.11)[13]. U człowieka enzym składa się z dwóch podjednostek. Mogą być to podjednostka α, β lub γ, z których składają się różne izoenzymu zawierające αα, αβ, αγ, ββ lub γγ[28][29]. Masa enzymu mieści w zakresie 82-100 kDa[28][30]. Podjednostki połączone są wiązaniem jonowym pomiędzy Glu-20 pierwszej podjednostki a Arg-414 drugiej podjednostki. Dla aktywności enzymu kluczowe są reszty His-159, Glu-168, Glu-211, Lys-345 i Lys-396[28]. Z centrum aktywnym połączone są dwa jony Mg2+ zapewniające stabilizację substratu oraz związku pośredniego w postaci karboanionu[29][28][31]. Dlatego Aktywność enzymu zależy od obecności jonów magnezu lub manganu[32][33], a hamowana jest w obecności fluorków. Działanie fluorków jest odpowiedzialne za zahamowanie aktywności enolazy bakterii odpowiedzialnych na wywoływanie próchnicy[34].
Synteza ATP 2

 | ADP ATP Kinaza pirogronianowa | 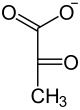 |
| Fosfoenolopirogronian | Pirogronian |
Przeniesienie grupy fosforanowej z PEP na ADP i powstanie ATP oraz pirogronianu katalizowane jest przez kinazę pirogronianową (PK, EC 2.7.1.40)[13]. Podczas przenoszenia grupy fosforanowej z enolofosforanu na ADP dochodzi do przekształcenia formy enolowej pirogronianu do stabilnej formy ketonowej[3]. Ze względu na znaczną utratę energii swobodnej w postaci ciepła, musi być traktowana jako reakcja fizjologicznie nieodwracalna. U większości badanych organizmów PK jest tetramerem zbudowanym z identycznych podjednostek. Enzym jest aktywny w obecności jonów K+ oraz Mg2+ i Mn2+. Różnice w budowie enzymu u różnych organizmów wiążą się ze zróżnicowaniem efektorów allosterycznych. Chociaż podstawowym związkiem regulującym aktywność enzymu jest fruktozo-2,6- fosforan to u wielu bakterii zwiększenie aktywności następuje pod wpływem monofosforanów cukrów takich jak 5-fosforan rybozy[36]. Dziedziczny niedobór kinazy pirogronianowej w erytrocytach może wywoływać niedokrwistość hemolityczną.
Substraty oddechowe
Podstawowym substratem dla glikolizy jest glukoza, jednak szlak jest podstawową drogi katabolizmu wielu heksoz. Fruktoza w organizmach zwierzęcych pochodzi z rozkładu sacharozy obecnej w pokarmie, a w organizmach roślinnych z rozkładu cukrów zapasowych takich jak sacharoza i inulina jest prowadzana do glikolizy na kilka sposobów[13][14]. W komórkach mięśni i tkance tłuszczowej fruktoza ulega fosforylacji przy udziale heksokinazy. W efekcie powstaje fruktozo-6-fosforan będący związkiem pośrednim w glikolizie. W komórkach wątroby podstawowym enzymem przeprowadzającym fosforylację glukozy jest glukokinaza. Włączenie fruktozy w glikolizę jest możliwe dzięki obecności dodatkowego enzymu, nazywanego fruktokinazą, przekształcającego fruktozę we fruktozo-1-fosforan, który jest rozszczepiany do aldehydu glicerynowego i fosfodihydroksyacetonu przez aldolazę fruktozo-1-fosforanu. Aldehyd glicerynowy ulega fosforylacji przeprowadzanej przez kinazę trioz. Dwa powstałe produkty są już metabolitami uczestniczącymi w glikolizie. Dwie dodatkowe reakcje określane są jako szlak fruktozo-1-fosforanowy[13]. W komórkach roślinnych fruktoza może być fosforylowana przez cytozolową heksokinazę lub fruktokinazę przyłączająca grupę fosforanową z ATP w pozycji 6[14].
Galaktoza, powstająca w organizmach zwierzęcych w wyniku rozkładu laktozy jest fosforylowana do glaktozo-1-fosforanu przez galaktokinazę. Następnie urydylilotransferaza galaktozo-1-fosforanowa przenosi grupę urydynową z UDP-glukozy wytwarzając UDP-galaktozę oraz glukozo-1-fosforan. UDP-galaktoza może być przekształcona do UDP-glukozy przez 4-epimerazę UDP-galaktozy. Opisane reakcje przekształcania galaktozy nazywane są szlakiem wzajemnych przemian galaktoza-glukoza. Powstający w drugiej reakcji szlaku glukozo-1-fosforan może być włączony w glikolizę poprzez przeniesienie grupy fosforanowej z pozycji 6 na pozycję 1 przez fosfoglukomutazę[13].
Dalsze losy pirogronianu
Powstający w wyniku zachodzenia reakcji glikolizy pirogronian może być przetworzony do różnych produktów anabolicznych. Najczęściej jednak jest katabolizowany poprzez utlenienie do acetylo-CoA, utlenianemu następnie w cyklu Krebsa. W warunkach beztlenowych pirogronian staje się substratem dla fermentacji[37].
Warunki tlenowe
U prokariontów w warunkach tlenowych pirogronian ulega utlenieniu i dekarboksylacji z udziałem kompleksu dehydrogenazy pirogronianowej[37].
W zdecydowanej większości ludzkich komórek (poza erytrocytami i włóknami mięśniowymi w czasie intensywnej pracy) przeważa metabolizm tlenowy. W warunkach tlenowych pirogronian, otrzymany w wyniku glikolizy pobierany jest przez mitochondria, w których po przekształceniu do acetylo-CoA (reakcja ta katalizowana jest przez wieloenzymatyczny kompleks dehydrogenazy pirogronianowej, do której działania niezbędna staje się difosfotiamina – pochodna witaminy B1), zostaje utleniony do dwutlenku węgla w cyklu Krebsa. Powstałe w glikolizie równoważniki redukujące są przenoszone zaś z NADH+H+ do wnętrza mitochondriów. Przenoszenie elektronów z cytoplazmatycznego NADH na mitochondrialny łańcuch oddechowy odbywa się dzięki krótkim sekwencjom reakcji zwanych tzw. czółenkami. Aktywne mogą być: czółenko glicerolo-3-fosforanowe lub jabłczanowo-asparaginianowe.
Arsenian, jony rtęciowe oraz niedobór tiaminy w diecie powodują nagromadzanie się pirogronianu w komórkach i doprowadzają do szybkiej kwasicy mleczanowej, będącej objawem niedoboru dehydrogenazy mleczanowej – prowadzi to u człowieka do zaburzeń neurologicznych poprzez zakłócenie podstawowych funkcji mózgu. Utlenieniu glukozy w warunkach tlenowych towarzyszy wytworzenie 38 moli ATP. Ogólny zysk wynosi więc 36 moli ATP[38].
Warunki beztlenowe
Jeśli przeważają warunki beztlenowe, niemożliwa staje się reoksydacja NADH w łańcuchu oddechowym przez przeniesienie równoważników redukujących na tlen. Pirogronian ulega redukcji przez NADH do mleczanu w reakcji katalizowanej przez dehydrogenazę mleczanową. Reoksydacja NADH w reakcji powstawania mleczanu przez odtworzenie NAD potrzebnego w następnym cyklu reakcji umożliwia dalszy przebieg glikolizy w przypadku nieobecności tlenu[38].
Przykładem komórek, które przeprowadzają wyłącznie glikolizę beztlenową są erytrocyty ssaków, ze względu na brak mitochondriów, niezbędnych do przeprowadzanie reakcji łańcucha oddechowego[38]. Jednak w przypadku krwinek czerwonych glikoliza zachodzi z ominięciem reakcji katalizowanej przez kinazę fosfoglicerynianową. Dodatkowy enzym, jakim jest mutaza bisfosfoglicerynianowa katalizuje przekształcenie 1,3-BPG w 2,3-BPG, który ostatecznie ulega przemianie do 3-fosfoglicerynianu przy udziale fosfatazy 2,3-bisfosfoglicerynianowej.
U prokariontów beztlenowych pirogronian może być przekształcony do acetylo-CoA w reakcja katalizowanej przez oksydazę pirogronian:ferredoksyna. Produktem reakcji poza acetylo-CoA i CO2 jest także zredukowana ferredoksyna. Inną możliwą droga przekształcenia pirogronianu jest reakcja katalizowana przez liazę pirogronian:mrówczan. Reakcja zachodzi u beztlenowych bakterii wydzielających mrówczan[37].
Możliwe jest także przekształcenie pirogronianu do aldehydu octowego przez dekarboksylazę pirogronianową. Aldehyd octowy jest następnie przekształcany do etanolu. Ta droga metabolizmu pirogronianu występuje u bakterii, drożdży i roślin[37][14]. Redukcja aldehydu przeprowadzana jest przez dehydrogenazę alkoholową przy użyciu NADH[14].
Organizmy roślinne przeprowadzają fermentację mleczanową w bardzo ograniczonym stopniu tylko przez krótki czas przy braku dostępu tlenu. Dłuższy okres anoksji prowadzi do rozpoczęcia procesów fermentacji alkoholowej[14].
Regulacja procesu glikolizy
Glikoliza regulowana jest na trzech etapach obejmujących z reakcji nieodwracalnych, to jest w miejscu działania heksokinazy (lub glukokinazy), fosfofruktokinazy I oraz kinazy pirogronianowej.
Regulacja heksokinazy
Reakcja katalizowana przez heksokinazę nie jest właściwa tylko dla glikolizy, gdyż jest to wspólny etap dla wszystkich dróg metabolizmu węglowodanów, w których glukoza jest substratem (szlak pentozofosforanowy, synteza glikogenu). Regulacja tej reakcji polega na hamującym działaniu glukozo-6-fosforanu. Związek ten gromadzi się gdy zahamowaniu ulega aktywność innego enzymu uczestniczącego w glikolizie – PFK. Glukozo-6-fosforan oraz fruktozo-6-fosforan pozostają w równowadze dzięki aktywności izomerazy glukozofosforanowej. Glukozo-6-fosforan może być jednak zużywany w komórkach zwierząt również do syntezy glikogenu, dlatego heksokinaza nie jest kluczowym enzymem w regulacji glikolizy[13].
Regulacja PFK
Najważniejszym miejscem regulacji glikolizy jest fosfofruktokinaza, czasami reakcja przeprowadzana przez ten enzym nazywana jest reakcją ograniczającą glikolizy. Aktywność enzymu regulowana jest na kilka sposobów. Pierwszy polega allosterycznym hamowaniu przez ATP. Efekt jest znoszony przez AMP. W ten sposób komórka może dostosować wytwarzanie ATP do aktualnego zapotrzebowania na energię. Gdy ilość ATP jest wystarczająca zachodzenie reakcji szlaku ulega ograniczeniu. Podwyższone stężenie AMP jest sygnałem do przyspieszenia glikolizy. Aktywność PFK ulega również zahamowaniu przy podwyższonych stężeniach cytrynianu, będącego jednym ze związków pośrednich cyklu Krebsa. Fruktozo-2,6-bisfosforan silnie aktywuje fosfofruktokinazę. Związek ten jest wytwarzany z fruktozo-6-fosforanu przez fosfofruktokinazę 2 (PFK 2) i może być hydrolizowany z powrotem przez fruktozobisfosfatazę (FBPazę). Ponieważ fruktozo-6-fosforan zwiększa aktywność PFK 2 i hamuje aktywność FBPazy to pośrednio wpływa na aktywność PFK. Fruktozobisfosfatazę zwiększa swoją aktywność również w efekcie fosforylacji, co ma miejsce w sytuacji podwyższonego stężenia ATP w cytozolu. Hydroliza fruktozo-2,6-bisfosforanu prowadzi do obniżenia aktywności PFK i spowolnienia całego szlaku glikolitycznego. Na aktywność PFK wpływają również jony H+. Do obniżenia pH cytozolu dochodzi, gdy w warunkach beztlenowych wytworzony zostanie kwas mlekowy[13].
Kinaza pirogronianowa
Kinaza pirogronianowa kontroluje wypływ metabolitów glikolizy. Jest ostatnią, nieodwracalną reakcją szlaku. Enzym aktywowany jest przez fruktozo-1,6-bisfosforan, a hamowany przez ATP oraz alaninę. W organizmach zwierząt dodatkowo kinaza pirogronianowa ulega fosforylacji przez kaskadę cAMP wywoływaną pojawieniem się glukagonu. Fosforylacja enzymu powoduje spowolnienie glikolizy i w efekcie podniesienie poziomu glukozy we krwi[13]. Allosteryczną regulację przez fruktozo-1,6-bisfosforan stwierdzono u ssaków. U mikroorganizmów w regulacji uczestniczą różne ufosforylowane cukry. Na przykład u świdrowców do aktywacji kinazy pirogronianowej dochodzi przy mikromolowych stężeniach fruktozo-2,6-bisfosforan[39].
Ewolucja
Glikoliza jako szlak metaboliczny służący pozyskiwaniu energii przydatnej metabolicznie wykształciła się na wczesnych etapach ewolucji. Reakcje szlaku umożliwiają pozyskanie energii z węglowodanów w warunkach beztlenowych[3]. Część szlaku, obejmująca metabolizm trioz jest obecna u organizmów należących do wszystkich trzech domen. Podobieństwo części enzymów odpowiedzialnych za metabolizm trioz wskazuje na ewolucyjne powiązanie glikolizy z glukoneogenezą[2]. Enzymy biorące udział w przekształcaniu heksoz różnią się u poszczególnych gatunków i nie występują wcale u części archeanów[40]. U archeanów występuje obok szlaku Embdena-Meyerhofa zmodyfikowany tryb glikolizy określany nazwą szlak Entnera-Doudoroffa[1][41]. Część glikolizy obejmująca metabolizm trioz, będąca zarazem częścią glukoneogenezy jest prawdopodobnie najstarszym ewolucyjnie fragmentem szlaku. Reakcje obejmujące przekształcanie heksoz wyewoluowały później,a różnice pomiędzy gatunkami mają związek dostępnością poszczególnych cukrów w środowisku organizmów. Badania sekwencji czterech kinaz i dwóch izomeraz uczestniczących w reakcjach glikolizy wskazują na odmienne pochodzenie tych enzymów. Nie powstały one w wyniku duplikacji i dywergencji jednego genu[3]. Długa ewolucja szlaku doprowadziła do wysokiej wydajności energetycznej w wyniku optymalizacji wytwarzania ATP. U części bakterii występują starsze ewolucyjnie szlaki glikolityczne o mniejszej wydajności[42].
Historia
Za początek badań nad glikolizą uznaje się obalenie poglądu Ludwika Pasteura z 1860 roku, że fermentacja jest procesem zachodzącym wyłącznie w żywych komórkach. Głoszony przez Pasteura dogmat witalistyczny obalili przypadkowo w roku 1897 bracia Hans i Eduard Buchnerowie, którzy starając się uzyskać ekstrakt drożdżowy do celów medycznych, próbowali utrwalić go za pomocą sacharozy. Okazało się, że pomimo braku żywych komórek sacharoza została szybko przekształcona w alkohol. W ten sposób udowodnili, że fermentacja może zachodzić poza żywą komórką. Przez kolejne lata poznano dokładnie reakcje fermentacji alkoholowej i mleczanowej. Udało się wykazać, że przebiegająca w mięśniach fermentacja mleczanowa jest w dużym stopniu podobna do fermentacji alkoholowej. Poznanie poszczególnych reakcji glikolizy było możliwe dzięki pracom Gustawa Embdena, Otto Meyerhofa, Carla Neuberga, Jakuba Parnasa, Otto Warburga, Gerty i Carla Corich. Wszystkie reakcje szlaku zostały poznane do roku 1940. Wkład części wymienionych naukowców został doceniony w stosowanej nazwie „szlaku Embdena-Meyerhofa-Parnasa”[3].
Galeria
Zobacz też
- metabolizm fruktozy
- glikogenogeneza
- glikogenoliza
- glukoneogeneza
Przypisy
- ↑ a b A.H. Romano, T. Conway, Evolution of carbohydrate metabolic pathways, „Research in Microbiology”, 147 (6-7), 1996, s. 448–455, DOI: 10.1016/0923-2508(96)83998-2, PMID: 9084754 [dostęp 2021-03-26] (ang.).
- ↑ a b Ron S. Ronimus, Hugh W. Morgan, Distribution and phylogenies of enzymes of the Embden-Meyerhof-Parnas pathway from archaea and hyperthermophilic bacteria support a gluconeogenic origin of metabolism, „Archaea”, 1 (3), 2003, s. 199–221, DOI: 10.1155/2003/162593, PMID: 15803666, PMCID: PMC2685568 [dostęp 2021-03-26] (ang.).
- ↑ a b c d e f g h i j k Glikoliza i glukoneogeneza, [w:] Jeremy M. Berg i inni, Biochemia, Warszawa: Wydawnictwo Naukowe PWN, 2007, s. 425–464, ISBN 978-83-01-14379-4.
- ↑ Jesús Muñoz-Bertomeu i inni, A critical role of plastidial glycolytic glyceraldehyde-3-phosphate dehydrogenase in the control of plant metabolism and development, „Plant Signaling & Behavior”, 5 (1), 2010, s. 67–69, DOI: 10.4161/psb.5.1.10200, PMID: 20592814, PMCID: PMC2835963 [dostęp 2021-03-26] (ang.).
- ↑ Vasilios M.E. Andriotis i inni, Plastidial glycolysis in developing Arabidopsis embryos, „The New Phytologist”, 185 (3), 2010, s. 649–662, DOI: 10.1111/j.1469-8137.2009.03113.x, PMID: 20002588 [dostęp 2021-03-26] (ang.).
- ↑ Wanderley de Souza, Special organelles of some pathogenic protozoa, „Parasitology Research”, 88 (12), 2002, s. 1013–1025, DOI: 10.1007/s00436-002-0696-2, PMID: 12444449 [dostęp 2021-03-26] (ang.).
- ↑ Jurgen R. Haanstra i inni, Compartmentation prevents a lethal turbo-explosion of glycolysis in trypanosomes, „Proceedings of the National Academy of Sciences of the United States of America”, 105 (46), 2008, s. 17718–17723, DOI: 10.1073/pnas.0806664105, PMID: 19008351, PMCID: PMC2584722 [dostęp 2021-03-26] (ang.).c?
- ↑ Marilyn Parsons, Glycosomes: parasites and the divergence of peroxisomal purpose, „Molecular Microbiology”, 53 (3), 2004, s. 717–724, DOI: 10.1111/j.1365-2958.2004.04203.x, PMID: 15255886 [dostęp 2021-03-26] (ang.).
- ↑ J. Maxwell Silverman i inni, Proteomic analysis of the secretome of Leishmania donovani, „Genome Biology”, 9 (2), 2008, R35, DOI: 10.1186/gb-2008-9-2-r35, PMID: 18282296, PMCID: PMC2374696 [dostęp 2021-03-26] (ang.).
- ↑ M. Parsons i inni, Biogenesis and function of peroxisomes and glycosomes, „Molecular and Biochemical Parasitology”, 115 (1), 2001, s. 19–28, DOI: 10.1016/s0166-6851(01)00261-4, PMID: 11377736 [dostęp 2021-03-26] (ang.).
- ↑ F.R. Opperdoes, Compartmentation of carbohydrate metabolism in trypanosomes, „Annual Review of Microbiology”, 41, 1987, s. 127–151, DOI: 10.1146/annurev.mi.41.100187.001015, PMID: 3120638 [dostęp 2021-03-26] (ang.).
- ↑ E. Bartholomeus Kuettner i inni, Crystal structure of hexokinase KlHxk1 of Kluyveromyces lactis: a molecular basis for understanding the control of yeast hexokinase functions via covalent modification and oligomerization, „Journal of Biological Chemistry”, 285 (52), 2010, s. 41019–41033, DOI: 10.1074/jbc.M110.185850, PMID: 20943665, PMCID: PMC3003401 [dostęp 2021-03-26] (ang.).c?
- ↑ a b c d e f g h i j k l m n o Metabolizm węglowodanów, [w:] B.D. Hames, N.M. Hooper, Krótkie wykłady. Biochemia, Warszawa: Wydawnictwo Naukowe PWN, 2005, s. 305–354, ISBN 83-01-13872-6.
- ↑ a b c d e f g Halina Gabryś. Procesy oddechowe, [w:] Jan Kopcewicz, Stanisław Lewak (red.), Fizjologia roślin, Warszawa: Wydawnictwo Naukowe PWN, 2002, s. 387–420, ISBN 83-01-13753-3.
- ↑ C.J. Jeffery i inni, Crystal structure of rabbit phosphoglucose isomerase, a glycolytic enzyme that moonlights as neuroleukin, autocrine motility factor, and differentiation mediator, „Biochemistry”, 39 (5), 2000, s. 955–964, DOI: 10.1021/bi991604m, PMID: 10653639 [dostęp 2021-03-26] (ang.).
- ↑ a b c B. Siebers i inni, Archaeal fructose-1,6-bisphosphate aldolases constitute a new family of archaeal type class I aldolase, „Journal of Biological Chemistry”, 276 (31), 2001, s. 28710–28718, DOI: 10.1074/jbc.M103447200, PMID: 11387336 [dostęp 2021-03-26] (ang.).c?
- ↑ E. Lorentzen i inni, Structure, function and evolution of the Archaeal class I fructose-1,6-bisphosphate aldolase, „Biochemical Society Transactions”, 32 (Pt 2), 2004, s. 259–263, DOI: 10.1042/bst0320259, PMID: 15046584 [dostęp 2021-03-26] (ang.).
- ↑ Nicola J. Patron, Matthew B. Rogers, Patrick J. Keeling, Gene replacement of fructose-1,6-bisphosphate aldolase supports the hypothesis of a single photosynthetic ancestor of chromalveolates, „Eukaryotic Cell”, 3 (5), 2004, s. 1169–1175, DOI: 10.1128/EC.3.5.1169-1175.2004, PMID: 15470245, PMCID: PMC522617 [dostęp 2021-03-26] (ang.).
- ↑ S.M. Zgiby i inni, Exploring substrate binding and discrimination in fructose1, 6-bisphosphate and tagatose 1,6-bisphosphate aldolases, „European Journal of Biochemistry”, 267 (6), 2000, s. 1858–1868, DOI: 10.1046/j.1432-1327.2000.01191.x, PMID: 10712619 [dostęp 2021-03-26] (ang.).
- ↑ H.C. Watson i inni, Sequence and structure of yeast phosphoglycerate kinase, „The EMBO Journal”, 1 (12), 1982, s. 1635–1640, PMID: 6765200, PMCID: PMC553262 [dostęp 2021-03-26] (ang.).
- ↑ G. Auerbach i inni, Closed structure of phosphoglycerate kinase from Thermotoga maritima reveals the catalytic mechanism and determinants of thermal stability, „Structure”, 5 (11), 1997, s. 1475–1483, DOI: 10.1016/s0969-2126(97)00297-9, PMID: 9384563 [dostęp 2021-03-26] (ang.).
- ↑ C.C. Blake, D.W. Rice, Phosphoglycerate kinase, „Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences”, 293 (1063), 1981, s. 93–104, DOI: 10.1098/rstb.1981.0063, PMID: 6115427 [dostęp 2021-03-26] (ang.).
- ↑ A.N. Szilágyi i inni, A 1.8 A resolution structure of pig muscle 3-phosphoglycerate kinase with bound MgADP and 3-phosphoglycerate in open conformation: new insight into the role of the nucleotide in domain closure, „Journal of Molecular Biology”, 306 (3), 2001, s. 499–511, DOI: 10.1006/jmbi.2000.4294, PMID: 11178909 [dostęp 2021-03-26] (ang.).
- ↑ R.D. Banks i inni, Sequence, structure and activity of phosphoglycerate kinase: a possible hinge-bending enzyme, „Nature”, 279 (5716), 1979, s. 773–777, DOI: 10.1038/279773a0, PMID: 450128 [dostęp 2021-03-26] (ang.).c?
- ↑ a b B.E. Bernstein, W.G. Hol, Crystal structures of substrates and products bound to the phosphoglycerate kinase active site reveal the catalytic mechanism, „Biochemistry”, 37 (13), 1998, s. 4429–4436, DOI: 10.1021/bi9724117, PMID: 9521762 [dostęp 2021-03-26] (ang.).
- ↑ S. Kumar i inni, Folding funnels and conformational transitions via hinge-bending motions, „Cell Biochemistry and Biophysics”, 31 (2), 1999, s. 141–164, DOI: 10.1007/BF02738169, PMID: 10593256 [dostęp 2021-03-26] (ang.).
- ↑ E. Zhang i inni, Mechanism of enolase: the crystal structure of asymmetric dimer enolase-2-phospho-D-glycerate/enolase-phosphoenolpyruvate at 2.0 Å resolution, „Biochemistry”, 36 (41), 1997, s. 12526–12534, DOI: 10.1021/bi9712450, PMID: 9376357 [dostęp 2021-03-26] (ang.).
- ↑ a b c d V. Pancholi, Multifunctional α-enolase: its role in diseases, „Cellular and Molecular Life Sciences”, 58 (7), 2001, s. 902–920, DOI: 10.1007/pl00000910, PMID: 11497239 [dostęp 2021-03-26] (ang.).
- ↑ a b M. Peshavaria, I.N. Day, Molecular structure of the human muscle-specific enolase gene (ENO3), „The Biochemical Journal”, 275 ( Pt 2), 1991, s. 427–433, DOI: 10.1042/bj2750427, PMID: 1840492, PMCID: PMC1150071 [dostęp 2021-03-26] (ang.).
- ↑ R.K.J. Hoorn, J.P. Flikweert, G.E.J. Staal, Purification and properties of enolase of human erythrocytes, „International Journal of Biochemistry”, 5 (11-12), 1974, s. 845–852, DOI: 10.1016/0020-711X(74)90119-0 [dostęp 2021-03-26] (ang.).
- ↑ T.M. Larsen i inni, A carboxylate oxygen of the substrate bridges the magnesium ions at the active site of enolase: structure of the yeast enzyme complexed with the equilibrium mixture of 2-phosphoglycerate and phosphoenolpyruvate at 1.8 Å resolution, „Biochemistry”, 35 (14), 1996, s. 4349–4358, DOI: 10.1021/bi952859c, PMID: 8605183 [dostęp 2021-03-26] (ang.).
- ↑ J.E. Wedekind, G.H. Reed, I. Rayment, Octahedral coordination at the high-affinity metal site in enolase: crystallographic analysis of the MgII–enzyme complex from yeast at 1.9 Å resolution, „Biochemistry”, 34 (13), 1995, s. 4325–4330, DOI: 10.1021/bi00013a022, PMID: 7703246 [dostęp 2021-03-26] (ang.).
- ↑ J.E. Wedekind i inni, Chelation of serine 39 to Mg2+ latches a gate at the active site of enolase: structure of the bis(Mg2+) complex of yeast enolase and the intermediate analog phosphonoacetohydroxamate at 2.1-Å resolution, „Biochemistry”, 33 (31), 1994, s. 9333–9342, DOI: 10.1021/bi00197a038, PMID: 8049235 [dostęp 2021-03-26] (ang.).
- ↑ F.J. Hüther, N. Psarros, H. Duschner, Isolation, characterization, and inhibition kinetics of enolase from Streptococcus rattus FA-1, „Infection and Immunity”, 58 (4), 1990, s. 1043–1047, DOI: 10.1128/IAI.58.4.1043-1047.1990, PMID: 2318530, PMCID: PMC258580 [dostęp 2021-03-26] (ang.).
- ↑ T.M. Larsen i inni, Structure of rabbit muscle pyruvate kinase complexed with Mn2+, K+, and pyruvate, „Biochemistry”, 33 (20), 1994, s. 6301–6309, DOI: 10.1021/bi00186a033, PMID: 8193145 [dostęp 2021-03-26] (ang.).
- ↑ G. Valentini i inni, The allosteric regulation of pyruvate kinase, „Journal of Biological Chemistry”, 275 (24), 2000, s. 18145–18152, DOI: 10.1074/jbc.M001870200, PMID: 10751408 [dostęp 2021-03-26] (ang.).c?
- ↑ a b c d Hans Günter Schlegel, Mikrobiologia ogólna, Zdzisław Markiewicz (red.), wyd. 2 popr., Warszawa: Wydawnictwo Naukowe PWN, 2004, s. 295–296, ISBN 83-01-13999-4, OCLC 749371403.
- ↑ a b c Peter A. Mayes, Glikoliza i utlenianie pirogronianu, [w:] Robert K. Murray i inni, Biochemia Harpera, Warszawa: Wydawnictwo Lekarskie PZWL, 1995, s. 207–216, ISBN 83-200-1798-X.
- ↑ William J. Cook i inni, Crystal structure of Cryptosporidium parvum pyruvate kinase, „PLoS One”, 7 (10), 2012, e46875, DOI: 10.1371/journal.pone.0046875, PMID: 23056503, PMCID: PMC3467265 [dostęp 2021-03-26] (ang.).c?
- ↑ T. Dandekar i inni, Pathway alignment: application to the comparative analysis of glycolytic enzymes, „The Biochemical Journal”, 343 Pt 1, 1999, s. 115–124, PMID: 10493919, PMCID: PMC1220531 [dostęp 2021-03-26] (ang.).
- ↑ M. Selig i inni, Comparative analysis of Embden-Meyerhof and Entner-Doudoroff glycolytic pathways in hyperthermophilic archaea and the bacterium Thermotoga, „Archives of Microbiology”, 167 (4), 1997, s. 217–232, DOI: 10.1007/BF03356097, PMID: 9075622 [dostęp 2021-03-26] (ang.).
- ↑ E. Meléndez-Hevia i inni, Theoretical approaches to the evolutionary optimization of glycolysis--chemical analysis, „European Journal of Biochemistry”, 244 (2), 1997, s. 527–543, DOI: 10.1111/j.1432-1033.1997.t01-1-00527.x, PMID: 9119021 [dostęp 2021-03-26] (ang.).
Linki zewnętrzne
- Pedro Jorge Araújo Alves da Silva, The chemical logic behind... Glycolysis, Universidade Fernando Pessoa [zarchiwizowane z adresu 2013-03-22] (ang.).
Media użyte na tej stronie
Autor: Thomas Splettstoesser (www.scistyle.com), Licencja: CC BY-SA 3.0
Visualization of the three domains of the protein Pyruvate kinase. Based on PDB: 1PKN
Part of the Biochem_reaction_arrow_Y_XXXX_horiz_med.png series for the metabolic pathways wikiproject.
Autor: Vossman, Licencja: CC BY-SA 3.0
Glyceraldehyde 3-phosphate dehydrogenase (GAPDH) with NAD+ and Pi bound to the active site. PDB rendering based on 2CZC.
Struktura alfa-D-glukozy (projekcja Hawortha)
Autor: Człowiek chrupek, Licencja: CC BY-SA 4.0
Schemat procesu glikolizy w oddychaniu tlenowym komórek wraz z modelami cząsteczek i ich uniwersalnymi nazwami
Autor: Zephyris z angielskiej Wikipedii, Licencja: CC-BY-SA-3.0
By Richard Wheeler (Zephyris) 2006. For the metabolic pathways wikiproject. en:Category:Protein images
Cartoon representation of the molecular structure of protein registered with 3pgk code.
Structure of dihydroxyacetonephosphate
arrow for chemical reactions, pointing right
Part of the Biochem_reaction_arrow_Y_XXXX_horiz_med.png series for the metabolic pathways wikiproject.
plus symbol (+), Times New Roman font at 400 px.
Structure of beta-D-fructose-1,6-bisphosphate
Autor:
- Hexokinase.pdb2.jpg: Jmol development team
- derivative work: Boghog (talk)
Hexokinase
Autor:
- Glikoliza.JPG: SkyMaja
- derivative work: Michał Sobkowski
Schemat glikolizy
Autor: Emw, Licencja: CC BY-SA 3.0
Structure of the GPI protein. Based on PyMOL rendering of PDB 1dqr.
Autor: Oryginalnym przesyłającym był Zephyris z angielskiej Wikipedii, Licencja: CC-BY-SA-3.0
By Richard Wheeler (Zephyris) 2006.
For the metabolic pathways wikiproject.Cartoon representation of the molecular structure of protein registered with 1fdj code.
transforming galatose (1) to glucose for the glycolysis. Galactose-1-phosphate (2), UDP-glucose (3), UDP-galatose (4) ; Glucose 1-phosphate (5); Glucose 6-phosphate (6). Galactokinase (GK), Galactose-1-phosphate uridylyltransferase (GALT), UDP-glucose 4-epimerase (UGE), phosphoglucomutase (PGM)
Structure of pyruvate in Fischer projection
Autor:
- Glycolysis2.svg: JohnyAbb
- derivative work: translated by Pisum
Glycolysis pathway overview. Updated file with minor corrections
Structure of beta-D-fructose-6-phosphate
Otto Fritz Meyerhof
Structure of D-glyceric aldehyde-3-phosphate
Structure of D-1,3-bisphosphoglycerate in Fischer projection
Structure of phosphoenolpyruvate in Fischer projection
(c) Zephyris z angielskojęzycznej Wikipedii, CC-BY-SA-3.0
By Richard Wheeler (Zephyris) 2006.
Structure of alpha-D-glucose-6-phosphate
Structure of alpha-D-fructose-6-phosphate
Autor: Dcrjsr, Licencja: CC BY 3.0
Ribbon schematic (hand drawn & colored) of the 3D structure of the protein triose phosphate isomerase. The barrel of 8 beta strands is shown by green arrows and the 8 alpha-helices as brown spirals. By Jane Richardson. [This white-background version (edited by Lycaon) less historically correct but probably more useful for pure illustration purposes.]
Structure of D-3-phosphoglycerate in Fischer projection
Structure of D-2-phosphoglycerate in Fischer projection