Interferencja RNA
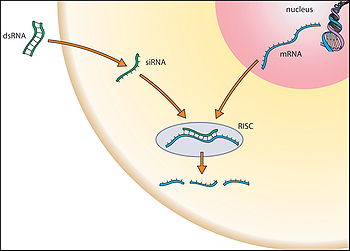
Interferencja RNA, RNAi (od ang. RNA interference) – zjawisko wyciszania albo wyłączenia ekspresji genu przez dwuniciowy RNA (dsRNA, od ang. double stranded RNA) o budowie i sekwencji zbliżonej do sekwencji DNA wyłączanego genu. Wyłączenie może się odbywać na trzech poziomach: a) degradacja mRNA; b) blokowanie translacji mRNA; c) prawdopodobnie również przez indukcję epigenetycznego wyciszenia genu.
Bezpośrednimi mediatorami interferencji RNA są małe,o długości 21-23 par zasad, dwuniciowe RNA, będące produktem obróbki większych fragmentów RNA przez specjalne nukleazy.
- Za degradację (zniszczenie) mRNA odpowiedzialne są oligonukleotydy tzw. małe interferujące RNA (siRNA, z ang. small interfering RNA), charakteryzują się stuprocentową homologią sekwencji do ich celu. Wchodzą w skład specjalnej rybonukleazy i nadają jej specyficzność do sekwencji. Mechanizm siRNA powstał najprawdopodobniej jako mechanizm obronny przed dwuniciowymi wirusami RNA (dsRNA). W konsekwencji degradacji mRNA odpowiedni gen jest wyciszany, bo nie powstaje kodowane przez niego białko.
- W przypadku mechanizmu interferencji z translacją mRNA, mediatorami są tzw. mikro-RNA (miRNA). miRNA kodowane są przez genom komórki, jak normalne geny. Prekursorem są niewielkie RNA, o strukturze spinki do włosów, które ulegają obróbce podobnie do siRNA. Wchodzą w skład kompleksów rybonukleoproteinowych blokujących specyficznie translację mRNA i nadają im specyficzność. W odróżnieniu od siRNA, miRNA nie posiadają 100%-owej homologii sekwencji do docelowego mRNA. miRNA są zaangażowane w negatywną regulację ekspresji genów podczas rozwoju.
Za odkrycie zjawiska interferencji RNA amerykańcy naukowcy Andrew Z. Fire i Craig C. Mello otrzymali w 2006 roku Nagrodę Nobla w dziedzinie medycyny i fizjologii. Dzięki badaniom i odkryciom noblistów i innych zespołów poznano fundamentalny mechanizm kontroli przepływu informacji genetycznej, które może mieć zastosowanie w terapii genowej. Już udało się wyciszyć gen odpowiedzialny za podwyższony poziom cholesterolu u zwierząt. W przyszłości będzie można opracować skuteczne metody leczenia chorób genetycznych i nowotworowych, a także nowe odmiany zwierząt i roślin hodowlanych.
Mechanizm
Małe interferujące RNA (siRNA) powstają przez pocięcie obcego dwuniciowego RNA (np. wirusowego) na krótkie kawałki (21-25 par zasad) przez białko Dicer, natomiast miRNA powstają jako długie cząsteczki prekursorowego miRNA transkrybowane z DNA komórki na RNA przez polimerazę RNA II (tę samą, która bierze udział w transkrypcji mRNA). Ocenia się, że miRNA biorą udział w regulacji 30% ludzkich genów. Następnie kompleks białkowy określany jako mikroprocesor rozpoznaje powstające w transkrypcie pre-miRNA struktury szpilki do włosów i tnie cząsteczkę na fragmenty o długości ok. 65 nukleotydów. Składają się one z dwuniciowego fragmentu o długości około 22 par zasad, na którego jednym końcu znajduje się jednoniciowa pętelka, a na drugim króciutki jednoniciowy kawałek. Taka szpilka RNA jest transportowana do cytoplazmy, gdzie Dicer przycina ją do odpowiedniej długości (likwidując też pętelkę).
Odpowiedniej długości miRNA i siRNA wiążą się z kompleksem białkowym o aktywności endorybonukleazy zwanym RISC (ang. RNA-induced silencing complex – indukowany przez RNA kompleks wyciszający). Cząsteczki RNA rozwijają się, a następnie kierują kompleks RISC do komplementarnych cząsteczek mRNA. siRNA są idealnie komplementarne do mRNA i w takim przypadku jeden ze składników RISC – białko Argonauta – przecina cząsteczkę mRNA. Jeśli cząsteczki nie wykazują się 100% homologią, jak to jest w przypadku miRNA, mRNA ulega strawieniu przez endorybonukleazy, lub następuje represja translacji tego mRNA. U roślin miRNA charakteryzują się zwykle wyższą homologią do docelowej sekwencji i indukują przecinanie mRNA przez RISC, a u zwierząt homologia miRNA do docelowego genu jest niższa, przez co powodują one represję translacji docelowego mRNA[2]. Wydaje się, że represja translacyjna polega na blokowaniu inicjacji translacji przez miRNA. Związanie się miRNA z docelowym transkryptem nie pozwala na łączenie się podjednostek rybosomu na tym mRNA, zapewne za pomocą białka eIF6[3]. Ponadto blokadzie inicjacji translacji ulegają tylko transkrypty, w których inicjacja translacji jest zależna od czapeczki, co sugeruje udział w tym mechanizmie czynnika inicjacji translacji eIF4E, który wiąże czapeczkę[4].
Białko Argonauta, miRNA i mRNA podlegające represji translacyjnej gromadzą się w ciałkach P. Tam odbywa się degradacja licznych mRNA oraz być może przechowywanie tych mRNA, które podlegają represji translacyjnej.
Historia odkrycia
Badania Napoli i wsp. nad petunią
Wszystko zaczęło się w 1990 roku na Uniwersytecie Arizony, gdzie grupa naukowców, w skład której wchodzili Carolyn Napoli, Christine Lemieux i Richard Jorgensen, planowała wyhodować odmianę petunii ogrodowej (Petunia hybryda) o kwiatach barwy ciemniejszej od odmiany pospolitej. Przeprowadzenie doświadczenia polegało na wprowadzeniu do komórek roślinnych dodatkowej kopii genu kodującego syntazę chalonową, enzym odpowiedzialny za syntezę fioletowego barwnika. W wyniku doświadczenia otrzymali petunię o kwiatach barwy jaśniejszej od pospolitej odmiany, zawierających białe plamy. Uzyskano kwiaty o barwie białej lub o częściowo białych kwiatkach. Wprowadzenie dodatkowego genu nie prowadziło do wydajniejszej syntezy barwnika, ale spowodowało zahamowanie wytwarzania barwnika kwiatu. W niewytłumaczalny i niezrozumiały wówczas sposób doszło do zahamowania syntezy barwnika, po wcześniejszym dodaniu kopii genu dla enzymu, którego funkcją jest jego synteza. Chcąc rozwikłać tę zagadkę, badacze zmierzyli w komórce roślinnej petunii poziom mRNA genu, którego dodatkową kopię dodali. Okazało się, że poziom mRNA tego genu spadł, co z jednej strony było zaskoczeniem, a z drugiej tłumaczyło to efekt doświadczenia. Obserwowane zjawisko miało tendencję do rozprzestrzeniania się po całym organizmie rośliny. W jaki zatem sposób doszło do wyciszenia ekspresji genu po dodaniu jego dodatkowej kopii? W komórkach rośliny istniał układ wyciszający i powodujący wyłączenie genu. Naukowcy zaobserwowany proces wyciszania genu nazwali kosupresją[5].
Badania Fire'a i Mello nad C.elegans
Przez 8 lat zagadka pozostawała niewyjaśniona, zjawisko pozostawało tajemnicą, aż do 19 lutego 1998 roku. Wytłumaczenie przyszło, gdy kolejna grupa naukowców, pracująca nad nicieniem Caenorhabditis elegans, odkryła, że wprowadzenie (iniekcja) dsRNA do komórek nicienia wycisza ekspresję genu, którego mRNA zawiera sekwencję komplementarną do wprowadzonego dsRNA, czyli wywołuje taki sam efekt, co wprowadzenie do komórek roślinnych dodatkowych kopii danego genu, a więc powoduje wyciszenie tego genu. Okazało się, że oba zjawiska mają taki sam mechanizm, a sygnałem uruchamiającym wyciszanie są właśnie cząsteczki dwuniciowego RNA. Wyciszanie genów za pomocą dwuniciowych cząsteczek RNA, czyli mechanizm mogący degradować mRNA dla specyficznego genu, nazwano interferencją RNA. W ten sposób dowiedziano się, że dsRNA jest kluczowym obiektem w mechanizmie wyciszania ekspresji genów. Mechanizm jest włączany, gdy w komórce pojawia się nietypowy, dwuniciowy RNA. „Dziwny” dwuniciowy RNA tworzy zwykłe „sensowy” mRNA w połączeniu z komplementarnym „antysensowym” RNA (czyli pasującym i uzupełniającym do mRNA). Andrew Z. Fire i Craig C. Mello wraz z zespołem opublikowali swoje odkrycie na łamach czasopisma Nature[6] i nazwali opisane zjawisko "interferencją RNA". Za to odkrycie w Andrew Z. Fire i Craig C. Mello otrzymali w 2006 roku Nagrodę Nobla w dziedzinie medycyny i fizjologii.
Przypisy
- ↑ R. Robinson. RNAi Therapeutics: How Likely, How Soon?. „PLoS Biol”. 2 (1), s. E28, 2004. DOI: 10.1371/journal.pbio.0020028. PMID: 14737201.
- ↑ A. Saumet, CH. Lecellier. Anti-viral RNA silencing: do we look like plants?. „Retrovirology”. 3, s. 3, 2006. DOI: 10.1186/1742-4690-3-3. PMID: 16409629.
- ↑ Chendrimada TP, Finn KJ, Ji X, Baillat D, Gregory RI, Liebhaber SA, Pasquinelli AE, Shiekhattar R. MicroRNA silencing through RISC recruitment of eIF6.. „Nature”. 447 (7146), s. 823-8, 2007. DOI: 10.1038/nature05841. PMID: 17507929.
- ↑ RJ. Jackson, N. Standart. How do microRNAs regulate gene expression?. „Sci STKE”. 2007 (367), s. re1, 2007. DOI: 10.1126/stke.3672007re1. PMID: 17200520.
- ↑ C. Napoli, C. Lemieux, R. Jorgensen. Introduction of a Chimeric Chalcone Synthase Gene into Petunia Results in Reversible Co-Suppression of Homologous Genes in trans.. „Plant Cell”. 2 (4), s. 279-289, 1990. DOI: 10.1105/tpc.2.4.279. PMID: 12354959.
- ↑ Andrew Fire, S.Q. Xu, Mary K. Montgomery, Steven A. Kostas, Samuel E. Driver, Craig C. Mello. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans.. „Nature”. 391 (6669), s. 806-11, 1998. DOI: 10.1038/35888. PMID: 9486653.
Linki zewnętrzne
- Katarzyna Kuleszewicz. Udział RNAi w wyciszaniu genów konkretnej tkanki, 16.01.2006
- Anna Olszewska. Strażnik genomu w służbie nauki, 07.10.2005
- M. Paprocka, M. Wołoszyńska. Potranskrypcyjne wyciszanie genów u roślin, 2004
- animacja z Nature przedstawiająca mechanizm zjawiska interferencji RNA
