Ligaza DNA
Ligaza DNA (EC 6.5.1.1) – enzym łączący wolne końce cząsteczek DNA.
Mechanizm działania
Łączenie DNA następuje poprzez wytworzenie wiązania fosfodiestrowego między końcem 3′ (hydroksylowym) a końcem 5′ (fosforowym) w reakcji zależnej od adenozynotrójfosforanu (ATP) w przypadku ligazy bakteriofagowej lub eukariotycznej albo od NAD+ w przypadku ligazy bakteryjnej. Reakcja katalizowana przez ligazę nazywa się ligacją.
Przykład działania ligazy (łączenie fragmentów DNA z końcami lepkimi):
z
5'-AGTCTGATCTGACT-3' 5'-pGATGCGTATGCTAGTGCT-3' 3'-TCAGACTAGACTGACTACGp-5' 3'-CATACGATCACGA-5'
powstaje
5'-AGTCTGATCTGACTGATGCGTATGCTAGTGCT-3' 3'-TCAGACTAGACTGACTACGCATACGATCACGA-5'
W tej reakcji tworzą się dwa rodzaje wiązań: wodorowe między nukleotydami komplementarnymi w obrębie lepkich końców (a właściwie ich zasadami azotowymi) i fosfodiestrowe pomiędzy pierwszym i ostatnim nukleotydem łączących się łańcuchów (a właściwie ich cząsteczkami deoksyrybozy). Wiązania wodorowe tworzą się spontanicznie, natomiast tworzenie wiązań fosfodiestrowych katalizuje właśnie ligaza DNA. Możliwa (choć mniej wydajna) jest też ligacja fragmentów zakończonych tępymi końcami. W tej reakcji powstają tylko nowe wiązania fosfodiestrowe.
Przykłady działania
- Podczas replikacji DNA na pewnym etapie nowa nić DNA składa się z osobnych części, tak zwanych fragmentów Okazaki. Za ich połączenie odpowiada ligaza.
- Uszkodzenia DNA w komórce doprowadzające do przerwania nici naprawia, między innymi, ligaza.
- Ligaza jest także jednym z kluczowych enzymów używanych w biologii molekularnej i inżynierii genetycznej.
Ligazy u różnych organizmów
U wszystkich bakterii występują ligazy zależne od NADH, a u niektórych (na przykład Haemophilus influenzae) również ligazy zależne od ATP. Te ostatnie wykazują się pewnym podobieństwem do ligaz bakteriofagów. Ligazy bakteriofagów i archeonów także są zależne od ATP, jednak homologia między nimi jest niewielka[1].
Ligazy organizmów eukariotycznych (oraz ich wirusów) są zależne od ATP. Ligaza DNA I bierze udział w łączeniu fragmentów Okazaki podczas replikacji DNA. Ligaza DNA II to fragment ligazy DNA III powstający w wyniku składania alternatywnego. Ligaza DNA III bierze udział w procesach naprawy DNA i rekombinacji. Ligaza DNA IV bierze udział w procesach naprawy DNA, rekombinacji niehomologicznej i w rekombinacji V(D)J[2].
Przypisy
- ↑ David J. Timson, Martin R. Singleton, Dale B. Wigley, DNA ligases in the repair and replication of DNA, „Mutation Research”, 460 (3-4), 2000, s. 301–18, DOI: 10.1016/S0921-8777(00)00033-1, PMID: 10946235.
- ↑ Ina V. Martin, Stuart A. MacNeill, ATP-dependent DNA ligases, „Genome Biology”, 3 (4), 2002, DOI: 10.1186/gb-2002-3-4-reviews3005, PMID: 11983065.
Linki zewnętrzne
- Molecule of the Month: DNA Ligase. nist.rcsb.org. [zarchiwizowane z tego adresu (2006-01-09)]., Protein Data Bank (ang.)
- Numer EC 6.5.1.1 w bazie Enzyme nomenclature database
Media użyte na tej stronie
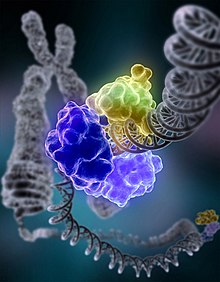
DNA damage, due to environmental factors and normal metabolic processes inside the cell, occurs at a rate of 1,000 to 1,000,000 molecular lesions per cell per day. A special enzyme, DNA ligase (shown here in color), encircles the double helix to repair a broken strand of DNA. DNA ligase is responsible for repairing the millions of DNA breaks generated during the normal course of a cell's life. Without molecules that can mend such breaks, cells can malfunction, die, or become cancerous. DNA ligases catalyse the crucial step of joining breaks in duplex DNA during DNA repair, replication and recombination, and require either Adenosine triphosphate (ATP) or Nicotinamide adenine dinucleotide (NAD+) as a cofactor.
Shown here is DNA ligase I repairing chromosomal damage. The three visable protein structures are:
- The DNA binding domain (DBD) which is bound to the DNA minor groove both upstream and downstream of the damaged area.
- The OB-fold domain (OBD) unwinds the DNA slightly over a span of six base pairs and is generally involved in nucleic acid binding.
- The Adenylation domain (AdD) contains enzymatically active residues that join the broken nucleotides together by catalyzing the formation of a phosphodiester bond between a phosphate and hydroxyl group.
