Modele budowy cząsteczek chemicznych

| Ten artykuł od 2021-02 wymaga zweryfikowania podanych informacji. |
Modele budowy cząsteczek chemicznych – fizyczne konstrukcje przedstawiające budowę cząsteczek chemicznych.
W najprostszych modelach atomy reprezentowane są przez kulki, połączone z sobą pręcikami, reprezentującymi wiązania. Jeśli średnice kulek będą proporcjonalne do promieni van der Waalsa atomów, a długości pałeczek do długości wiązań, to otrzymamy model mniej więcej zgodny z rzeczywistą budową cząsteczki.
W rzeczywistości długości i kąty wiązań chemicznych zmieniają się w bardzo szerokim zakresie w różnych cząsteczkach i dlatego tego rodzaju modele nie odzwierciedlają w pełni budowy cząsteczek.
Określeniem struktury danej cząsteczki poprzez analizę oddziaływania jej składników zajmuje się modelowanie molekularne.
Techniki modelowania budowy cząsteczek

Chemia teoretyczna rozwinęła szereg metod obliczeniowych pozwalających na mniej lub bardziej precyzyjne ustalanie faktycznej struktury przestrzennej cząsteczek.
Metody te można podzielić na:
- mechanikę molekularną – metody te traktują atomy jak kule o określonej średnicy i masie, a wiązania jak sprężyny, których sposób rozciągania się i zginania odpowiada mniej więcej własnościom wiązań chemicznych. Parametry owych kul i sprężyn ustala się na podstawie analizy związków chemicznych, których struktura jest znana np. z rentgenografii strukturalnej. Metody te nie wymagają dużych mocy obliczeniowych, mogą więc być stosowane do nawet bardzo dużych cząsteczek, są one jednak tylko nieco dokładniejsze od modeli prymitywnych i zupełnie zawodzą dla nietypowych cząsteczek.
- metody półempiryczne – metody te bazują już na równaniu Schrödingera: dla danej cząsteczki tworzony jest układ liniowych równań różniczkowych, przy czym wszystkie parametry tych równań są ustalane na podstawie struktury znanych związków. Następnie układ taki jest rozwiązywany metodą kolejnych przybliżeń, aż do znalezienia minimum energetycznego dla modelowanej cząsteczki. Metody te są znacznie bardziej dokładne od mechaniki molekularnej, ale podobnie jak mechanika działają one poprawnie tylko dla w miarę typowych związków chemicznych i zupełnie zawodzą przy nietypowych strukturach.
- metody ab initio.
Oprócz tego istnieją też metody umożliwiające symulowanie ruchu cząsteczek i oddziaływań między nimi zwane dynamiką molekularną.
Sposoby przedstawiania modeli
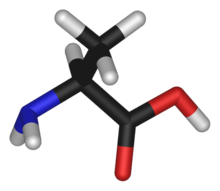

Modele cząsteczek tradycyjnie przedstawia się na trzy zasadnicze sposoby:
- Model kulkowy – w którym obrazowane są połączenia między atomami z pominięciem ich promieni atomowych. Modele takie dobrze uwidaczniają strukturę geometryczną połączeń między atomami, mogą jednak tworzyć błędne wrażenie co do rzeczywistej objętości zajmowanej przez cząsteczki i ich ogólnego zewnętrznego kształtu.
- Model czaszowy – w którym przedstawia się możliwie realistycznie atomy jako kule o średnicach zbliżonych do ich rzeczywistych promieni van der Waalsa – oczywiście powiększonych do skali modelu. W modelach takich nie widać szkieletu wiązań, ale za to lepiej obrazują one ogólny kształt cząsteczki.
- Model pręcikowo-kulkowy – w którym promienie chmur elektronowych atomów są w stosunku do siebie proporcjonalne, ale są one wszystkie podzielone przez pewien współczynnik, dzięki czemu widać w tych modelach także układ wiązań.
Wszystkie sposoby przedstawiania modeli cząsteczek są daleko idącym uproszczeniem schematycznym, rodzajem umownej konwencji ich przedstawiania. Ich rzeczywista struktura elektronowa i własności są zupełnie inne niż można by oczekiwać od zestawu twardych kulek połączonych prętami. Cząsteczki są bowiem obiektami, które opisuje się przy pomocy pojęć chemii kwantowej i które w istocie nie zachowują się tak jak obiekty makroskopowe znane z codziennego życia.

Media użyte na tej stronie
Autor: Effeietsanders, Licencja: CC BY-SA 2.5
Cafeïne-molecuul. Rood = zuurstof, lichtblau = koolstof, donkerblauw = stikstof, wit = waterstof
Autor:
- derivative work: Dhatfield (talk)
- MM_PEF.svg: Edboas
3D render of a molecular model. The molecule is alanine peptide. The shroud represents the region of hydrophobic repulsion, where the strength of the hydrophobic effect is approximately proportional to the surface area of the shroud. The shroud, shown extending only over the back of the molecule extends all the way around it. This diagram can be used to demonstrate the variation in potential energy as bonds shift either in rotation, angle or translation, as seen in the image MM_PEF_3.png.
glucose molecule
Autor: Peter Murray-Rust, Licencja: CC BY-SA 2.5
molecular model of small peptide