Ostatni uniwersalny wspólny przodek

Ostatni uniwersalny wspólny przodek (ang. last universal common ancestor, LUCA), ostatni uniwersalny przodek (ang. last universal ancestor, LUA) – hipotetyczny organizm, który był ostatnim wspólnym przodkiem wszystkich żyjących obecnie na Ziemi, należących do domeny bakterii, archeanów i eukariontów[1][2]. Pogląd o istnieniu organizmu, który dał początek wszystkim występującym obecnie organizmom jest szeroko rozpowszechniony wśród biologów.
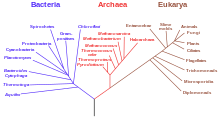
Natura wspólnego przodka może być poznana jedynie poprzez molekularne badania struktury i sekwencji elementów współczesnych organizmów. Efektem analiz jest stworzenie drzewa życia, czyli drzewa filogenetycznego obejmującego wszystkie trzy domeny[2].
Według jednych hipotez taki pierwotny organizm miał budowę i cechy zbliżone do bakterii hipertermofilnych, jednak wiele nowych danych wskazuje że LUCA był termofilnym lub mezofilnym proteoeukariontem, od którego pochodzą współczesne eukarionty, a bakterie i archeany pojawiły się na jego wczesnych etapach ewolucji[3][4][5]. Pochodzący od progenotów proteoeukariont mógł zawierać DNA, jednak za prawdopodobne uznaje się, że genom tego organizmu był zbudowany z RNA, a zmiana na DNA nastąpiła dopiero po wykształceniu trzech domen[5].
Rozwój hipotezy

Hipoteza zakładająca istnienie uniwersalnego wspólnego przodka wszystkich organizmów jest podstawą współczesnej teorii ewolucji. Została przedstawiona już przez Darwina w dziele „O powstawaniu gatunków”. Dotychczas nie udało się empirycznie rozstrzygnąć, czy życie na Ziemi jest całkowicie monofiletyczne, jeden gatunek dał początek wszystkim, czy też, jak sugerują niektórzy naukowcy, na wczesnym etapie rozwoju życia poziomy transfer genów był na tyle powszechny, że monofiletyzm życia nie jest możliwy[7]. Niezależnie od tego, czy LUCA był jednym gatunkiem, czy współpracującą ze sobą grupą organizmów, jego istnienie datowane jest na 3,5-3,8 mld lat temu[6]. Najbardziej podstawowy podział na trzy domeny wywodzące się od wspólnego przodka zaproponowali Fox i Woese w roku 1977 na podstawie sekwencji rRNA[8]. Kolejne badania molekularne doprowadziły do wniosku, że jako pierwsze oddzieliły się bakterie. Następnie ze wspólnej linii powstały eukarionty i archeany. Jądro komórkowe eukariontów mogło powstać w wyniku wbudowania w komórkę kompleksu wirusów DNA[9][10]. W 2010 roku oszacowano, że hipoteza o pochodzeniu wszystkich obecnie żyjących organizmów od jednego gatunku była 102830 razy bardziej prawdopodobna niż najlepsza alternatywna hipoteza[7].
Genom i proteom
Większość szacunków uznaje, że genom LUCA musiał zawierać co najmniej 500-1000 genów[11]. Założenie, że hipotetyczny genom przodka wszystkich domen powinien być mały i zbliżony do prostych organizmów pasożytniczych, nie jest jednak poparte dowodami. Liczba rodzin genów u prokariontów mieści się w granicach 1006 do 1189, a u eukariontów pomiędzy 1344 a 1529. Bardziej prawdopodobne jest, że genom LUCA zawierał liczbę genów porównywalną z wolno żyjącymi współczesnymi prokariotami[12]. Zidentyfikowano co najmniej 140 domen białek tworzących prawdopodobnie proteom LUCA[13]. Rekonstrukcja sekwencji domen białkowych wskazuje, że charakteryzowały się one wysoką hydrofobowością[14].
Przypisy
- ↑ C.R. Woese, O. Kandler, M.L. Wheelis, Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya, „Proceedings of the National Academy of Sciences of the United States of America”, 87 (12), 1990, s. 4576–4579, DOI: 10.1073/pnas.87.12.4576, PMID: 2112744, PMCID: PMC54159.c?
- ↑ a b C. Woese, The universal ancestor, „Proceedings of the National Academy of Sciences of the United States of America”, 95 (12), 1998, s. 6854–6859, DOI: 10.1073/pnas.95.12.6854, PMID: 9618502, PMCID: PMC22660.c?
- ↑ N. Glansdorff, About the last common ancestor, the universal life-tree and lateral gene transfer: a reappraisal, „Molecular Microbiology”, 38 (2), 2000, s. 177–185, DOI: 10.1046/j.1365-2958.2000.02126.x, PMID: 11069646.
- ↑ Ying Xu, Nicolas Glansdorff, Was our ancestor a hyperthermophilic procaryote?, „Comparative Biochemistry and Physiology. Part A, Molecular & Integrative Physiology”, 133 (3), 2002, s. 677–688, DOI: 10.1016/S1095-6433(02)00197-6, PMID: 12443925.
- ↑ a b Nicolas Glansdorff, Ying Xu, Bernard Labedan, The last universal common ancestor: emergence, constitution and genetic legacy of an elusive forerunner, „Biology Direct”, 3, 2008, art. nr 29, DOI: 10.1186/1745-6150-3-29, PMID: 18613974, PMCID: PMC2478661.
- ↑ a b c W.F. Doolittle, Uprooting the tree of life, „Scientific American”, 282 (2), 2000, s. 90–95, PMID: 10710791 [dostęp 2019-03-24].c?
- ↑ a b Douglas L. Theobald, A formal test of the theory of universal common ancestry, „Nature”, 465 (7295), 2010, s. 219–222, DOI: 10.1038/nature09014, PMID: 20463738.c?
- ↑ M.F. White, Archaeal DNA repair: paradigms and puzzles, „Biochemical Society Transactions”, 31 (Pt 3), 2003, s. 690–693, DOI: 10.1042/bst0310690, PMID: 12773184.
- ↑ P.J. Bell, Viral eukaryogenesis: was the ancestor of the nucleus a complex DNA virus?, „Journal of Molecular Evolution”, 53 (3), 2001, s. 251–256, DOI: 10.1007/s002390010215, PMID: 11523012.
- ↑ Philip John Livingstone Bell, The viral eukaryogenesis hypothesis: a key role for viruses in the emergence of eukaryotes from a prokaryotic world environment, „Annals of the New York Academy of Sciences”, 1178, 2009, s. 91–105, DOI: 10.1111/j.1749-6632.2009.04994.x, PMID: 19845630.
- ↑ Arcady Mushegian, Gene content of LUCA, the last universal common ancestor, „Frontiers in Bioscience”, 13, 2008, s. 4657–4666, DOI: 10.2741/3031, PMID: 18508537.
- ↑ Christos A. Ouzounis i inni, A minimal estimate for the gene content of the last universal common ancestor--exobiology from a terrestrial perspective, „Research in Microbiology”, 157 (1), 2006, s. 57–68, DOI: 10.1016/j.resmic.2005.06.015, PMID: 16431085.
- ↑ Juan A.G. Ranea i inni, Protein superfamily evolution and the last universal common ancestor (LUCA), „Journal of Molecular Evolution”, 63 (4), 2006, s. 513–525, DOI: 10.1007/s00239-005-0289-7, PMID: 17021929.
- ↑ Ranjan V. Mannige, Charles L. Brooks, Eugene I. Shakhnovich, A universal trend among proteomes indicates an oily last common ancestor, „PLoS Computational Biology”, 8 (12), 2012, art. nr e1002839, DOI: 10.1371/journal.pcbi.1002839, PMID: 23300421, PMCID: PMC3531291.
Linki zewnętrzne
- Douglas Theobald: 29+ Evidences for Macroevolution: The Scientific Case for Common Descent. www.talkorigins.org, 1999-2012. [dostęp 2014-06-25].
| ||||||||||||||||||||||||||
Media użyte na tej stronie
Darwin's finches or Galapagos finches. Darwin, 1845. Journal of researches into the natural history and geology of the countries visited during the voyage of H.M.S. Beagle round the world, under the Command of Capt. Fitz Roy, R.N. 2d edition.
- 1. (category) Geospiza magnirostris
- 2. (category) Geospiza fortis
- 3. Geospiza parvula, now (category) Camarhynchus parvulus
- 4. (category) Certhidea olivacea
A phylogenetic tree of living things, based on RNA data and proposed by Carl Woese, showing the separation of bacteria, archaea, and eukaryotes. Trees constructed with other genes are generally similar, although they may place some early-branching groups very differently, thanks to long branch attraction. The exact relationships of the three domains are still being debated, as is the position of the root of the tree. It has also been suggested that due to lateral gene transfer, a tree may not be the best representation of the genetic relationships of all organisms. For instance some genetic evidence suggests that eukaryotes evolved from the union of some bacteria and archaea (one becoming an organelle and the other the main cell).
Autor: Origionally NASA, Licencja: CC-BY-SA-3.0
Modified version of Image:PhylogeneticTree.png showing horizontal gene transfer events, based on http://www.nature.com/nrmicro/journal/v3/n9/fig_tab/nrmicro1253_F1.html